
저의 대학원생활의 첫 질량분석기는 지금은 볼수 없는 Agilent사의 iontrap이 었습니다. 그 당시 Thermo Finnigan 사의 LCQ 나 LTQ가 가장 좋은 질량분석기였습니다. 몇년후 LTQ를 구매하고 나서 Nature 급 논문을 낼 수 있을것이라 생각했으나 이미 그 시대는 지났습니다. Oribtrap 의 첫 모델이 나왔기 때문입니다. Orbitrap을 구매했을 때는 Q-Exactive 가 다시 대세였습니다.
미국에 왔을때 실험실에 Orbitrap Velos가 있었습니다. 나머지는 모두 Q-Exacdtive 와 Q-Exacdtive plus 였습니다. 실험자체가 PRM 위주였기도 하였지만 아무도 Oribrap Velos를 사용하지 않았습니다. 그냥 컬럼테스트용도 였습니다.
Orbitrap 초기모델에 익숙한 저에게는 Oritrap Velos는 감지덕지한 장비였습니다. 비록 감도가 다른 장비에 비해 낮더라도 긴 컬럼과 긴 LC time을 사용하면 낮은 감도를 어느정도 보완이 가능합니다. 그래서 장비는 혼자 점령하고 계속 분석하는것은 여러모로 장점이 있습니다. 바로 시간이 많다는 것이지요.
다행히 거의 혼자 독점하다시피 실험할수 있어서 짧은 기간에 논문을 한개 낼 수 있었습니다.
UPENN에서 일을 시작할때 Q-Exactive가 Q-Exactdive HF, HFX, Orbitrap Fusion에 밀려서 찬밥시세 였습니다. 그리고 최근에 우선순위가 떨어지는 프로젝트를 위해 상대적으로 저가인 Q-Exactive를 구매했습니다.
아래는 새 Q-Exactive가 설치된후 장비성능을 테스트 한 결과들입니다. 결론은 비로 Old fasion Q-Exactive이지만 새 장비는 어느정도 사용한 Q-Exactive HF 나 HFX 만큼의 검출감도가 나왔습니다.
Mass spectrometry: Q-Exactive
▪ nanoLC: Easy nLC 1000
▪ Samples: Standard BSA (100fmol), HeLa (200ng on Column, P/N 88328 Pierce)
LC Gradient 최적화
테스트를 진행할때는 LC gradient 조건을 가능한 최적화 한 상태에서 하는것이 좋습니다. 일단 어느정도 좋은 크로마토그램을 얻게 되면 테스트 과정에서 단백질 혹은 펩타이드의 수가 예상보다 적게 검출시 LC 에서 발생하는 문제가 아님을 확신할수 있습니다. 즉 LC troubleshooting 에 필요없이 시간을 낭비하지 않습니다.
아래 크로마토 그램은 200 ng HeLa digest standard를 60 min LC gradient로 분리한것입니다. 펩타이드가 분리되는 구간은 전체 60 min에서 3분에서 54분으로 아주 넓게 퍼져있습니다. 특별히 낭비되는 시간없이 피크들이 알차게 퍼져있습니다.

120 min gradient에서도 동일하게 펩타이드들이 3~110 min 사이에 골고루 퍼져있습니다.

검출된 단백질 및 펩타이드 수
아래 그래프는 200ng HeLa standard digest를 각각의 LC gradient에서 측정한 데이타입니다. 1hr Gradient에서 약 3,000개의 단백질이 검출되었습니다(Proteome discoverer D2.4, 1% FDR). Q-Exactive HF 와 HFX의 경우도 유사한 조건에서도 대략 3,000개 정도의 단백질이 검출되었으니 꽤 좋네요.

Isolation Window (m/z)
LTQ 나 초기 Orbitrap에서 QE 시리즈로 넘어갈때 향상된 부분중 하나가 ion isolation efficiency 입니다. Iontrap이 아닌 Quadrupole을 이용하기 때문이겠지요. Iontrap에서 사용하던 2.0 mz보다는 조금더 낮은 값을 사용해도 될것같습니다. 이론적으로 말하자만, 좁은 isolation을 사용하게 되면 이웃한 피크가 같이 fragmentation 될수 있는 기회를 줄일수 있게 되어 조금더 좋은 MS2 스펙트럼을 얻을수 있습니다. 또한 다른 피크들이 제거 됨으로서 상대적은 이온들의 수가 낮아지게 됨으로 같은 ion accumulation에서 보다 많은 이온을 얻을수 있습니다. 다른 펩타이의 이온이 포함된 1000개의 이온과 순수한 펩타이드만 포함된 1000개 이온에 대한 스펙트럼은 아마 다를것입니다.
아래 그래프는 동일한 조건하에 isolation window 차이에 따른 펩타이드 검출수를 나타낸다. 하지만 검출된 펩타이드의 수는 그다지 차이가 나지 않네요. 1.5 m/z 가 그나마 좋은것 같습니다.

다른 isolation에서 검출된 Actin 의 대표적인 펩타이드의 MSMS 스펙트럼을 보겠습니다.
아래는 QEYDESGPSIVHR 에 대한MS2 스펙트럼입니다. Isolation window가 좁아 질 수록 MS2 의 product ion 수도 감소되는 경향이 있습니다. 1.0 m/z에서는 검출이 되지 않았습니다.

Actin의 또 다른 펩타이드인 SYELPDGQVITIGNER 의 경우도 아래와 같이 유사한 패턴을 보입니다.
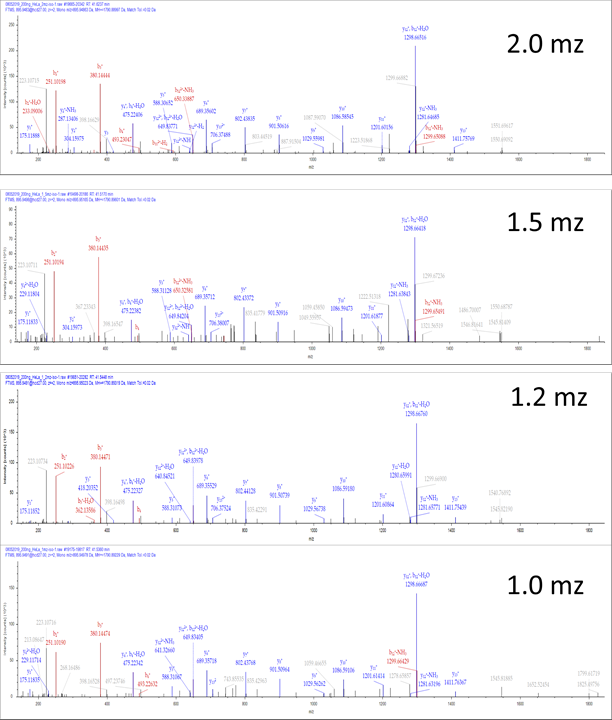
검출된 펩타이드의 수와 MS2 스펙트럼의 경향을 보아서는 대략 1.5 m/z가 좋아보입니다. 하지만 이것도 역시 시료에 따라 그리고 장비의 calibration 여부에 따라 약간씩 달라지겠지요. 그래도 아무런 정보가 없으면 일단 1.5m/z 정도에서 시작하는것이 좋겠네요.
Intensity Threshold
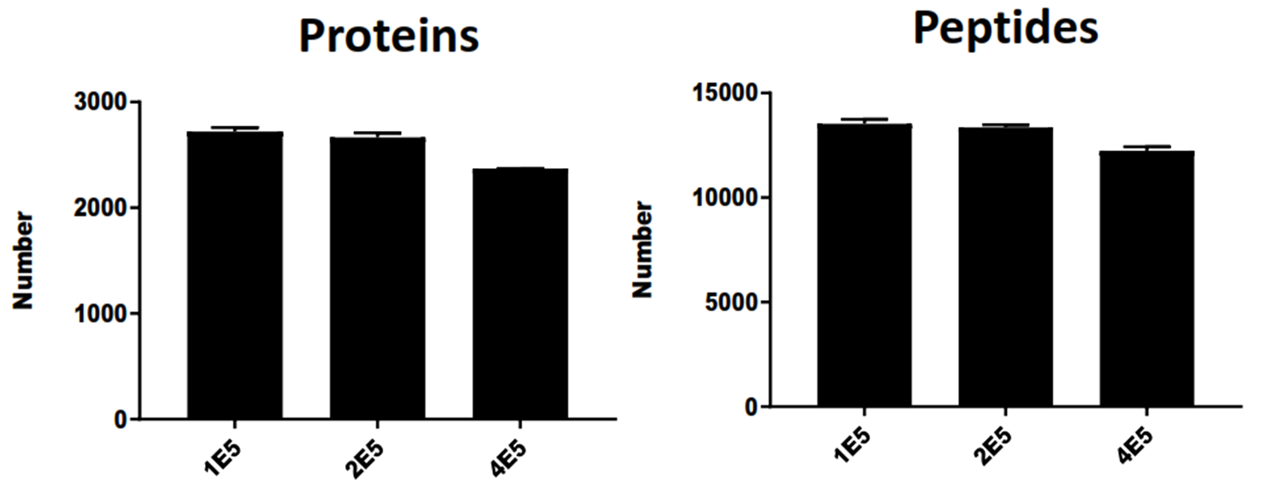
이 파라미터는 그다지 중요하지 않은것 같습니다 하지만 어떤 영향이 있는지 확인하고자 간단히 테스트 해보았습니다. Intensity Threshold는 말 그대로 정해진 Threshold 이상의 MS1 피크만을 MS2를 수행하는것입니다. 너무 높으면 낮은 피크들이 검출될 가능성이 줄어들게 되는 반면 너무 낮게 되면 MS1 피크가 충분히 높지 않은 상태에서 MS2를 수행할수 있기 때문에 MS2 스펙트럼의 질이 떨어질 가능성이 있습니다. 하지만 보통 DDA에서는 Intensity가 높은 순으로 MS2를 수행하기 때문에 Threshold값에 따라 검출되는 펩타이드의 수가 확연히 차이가 나지는 않을 것입니다. 아래는 200 ng HeLa standard digest를 이용하여 default 값 근처의 3가지 범위에서 얻어진 단백질 및 펩타이드 수 입니다. Threshold가 1E5, 2E5, 4E5로 갈수록 검출되는 펩타이드의 수가 조금씩 감소하는 경향을 보입니다. 아마 Threshold 보다 약간 낮은 intensity를 가지는 펩타이드들이 검출되지 않았기 때문일것입니다. 하지만 시료의 양이 많은 경우와 적은 경우에 따라 Threshold를 적절히 바꾸어 주면 좋은 MS2를 얻을수 있을 뿐만 아니라 검출되는 단백질 및 펩타이드의 수가 동시에 증가 시킬수 있을 것입니다.
Q-Exactive의 accuracy 는 그다지 변동이 심하지 않지만 그래도 시간이 지남에 따라 ppm값이 변하게 됩니다. MS calibration으로 보정하면 되지만 시료분석시 internal calibration을 위해서 Lock Mass option을 사용하면 일정 기간동안은 따로 calibration이 없이 아주 낮은 ppm값으로 검출이 가능합니다.
일반적으로 background 피크에서 발견된는 Polysiloxane 인 445.1200 ([C2H6SiO]6) 혹은 371.10124([C2H6SiO]5)를 사용합니다. 두 peak중 높은 intensity를 가지는 것을 하나 선택해서 사용하면 됩니다. 초기 Orbitrap 장비의 Lock mass option을 사용하면 scan time에 영향을 주어 검출되는 단백질/펩타이드 수가 감소 할수 있다는 말이 있었습니다. 하지만 아래 그래프에서 보듯이 QE의 경우 오히려 검출된 단백질과 펩타이드의 수가 아주 약간 증가하였습니다. Lock mass 는 그냥 사용하는것이 좋을것 같습니다.

'프로테오믹스(단백체학)' 카테고리의 다른 글
| p 값에 대해 간단한 해석 (0) | 2020.06.22 |
|---|---|
| Formic acid(F.A) 와 trifluoroacetic acid (TFA)의 Trap-column에 대한 펩타이드 결합력 비교 (0) | 2020.06.21 |
| TMT 정량의 분석에서의 SPS MS3 방법은 진짜 좋은가? (1) | 2020.06.12 |
| Web-based tool normalize omics data: Normalizer 사용법 (0) | 2020.06.07 |
| BioPureSPN HILIC (ERLIC) Spin-column의 용량및 성능검증 (0) | 2020.06.03 |



댓글