Bruker사의 timsTOF 가 출시되고 관심을 받는 시기에 FAIMS를 접했던터라 FAIMS도 timsTOF과 동일한 기능을 가진것이라고 생각했습니다. LC에서 분리된 펩타이드를 추가적으로 분리한다는 점에서는 동일한 목적을 가지고 있지만 분리되는 메카니즘은 다른것 같습니다.
설치위치
FAIMS는 Orbitrap장비의 외부에 장착됩니다. Nano & ESI source가 장착되는 부분에 FAIMS 를 장착합니다. 그리고 다시 그 앞에 nano ESI를 장착합니다. LC에서 분리된 펩타이들이 FAIMS를 거친후 선택적으로 질량분석기로 들어갑니다.

처음 FAIMS를 접했을때 Bruker사의 IMS와 같은 역활을 기대했습니다. 물론 두 시스템에 대해서 전혀 몰랐을때 입니다 (사실 지금도 잘 모릅니다.) FAIMS는 장비 밖에서 붙히고 뗄수 있지만 timsTOF의 ion mobility는 질량분석기 안에 설치되어 있어서 분리가 안됩니다.
FAIMS 에는 Compensation voltage (CV)를 조절해서 펩타이드를 분리시킵니다. HPLC에서 분리되는것과는 다릅니다. LC에서 분리된 펩타이드들이 CV값에 따FAIMS에서 빠져나와서 질량분석기속으로 들어갈수도 있고 아니면 FAIMS안에서 그냥 붙어버리릴수도 있습니다. 서로 다른 CV값에 따라 안정된 펩타이드들의 종류가 다르기 때문에 서로 다른 CV 값을 적용하면 그때 마다 서로 다른 펩타이드들이 질량분석기로 들어갑니다. LC에서 빠져나온 펩타이드들이 한꺼번에 질량분석기로 들어가지 않고 나누어서 들어가게 됩니다.
일단 FAIMS를 장착한후 사용하고 싶지 않을때 FAIMS를 떼어내어야 합니다. FAIMS를 장착한 상태에서 Method에서 OFF를 하여서 사용하지 않습니다. OFF를 해서 사용할수 있지만 그렇게 할 이유가 없습니다. 이온들이 굳이 사용하지 않는 FAIMS를 통과해서 질량분석기를 통과하게 할 필요가 없습니다. 감도가 떨어지겠지요.
FAIMS를 장착하여 서로 다른 CV값을 적용하면 감도가 증가되는 이유는 대략 아래와 같을듯합니다.
1)Reduce background peaks
FAIMS에서 펩타이드들이 안정한 CV값에서는 대부분의 background peak들이 불안정하여 제거됩니다. FAIMS를 사용하면 TIC값이 상당히 낮아지는 이유입니다. 일반적으로 보여지 background peak가 FAIMS 상태에서는 상당히 낮거나 검출되지 않습니다. 질량분석기는 background 피크나나 펩타이드 피크나 구별하지 않고 들어오는 이온화된 모든 이온들을 다 받아들입니다. 하지만 질량분석기안에서 모아둘수 있는 이온들의 공간은 제한적입니다. 그러므로 background 피크에 해당하는 이온들 질량분석기 안에 들어오기전에 제거할수있다면 펩타이드 이온들을 더 많이 모을수 있게 됩니다.
2)Reduce peptide complexity
질량분석 스펙트럼을 보면 왼쪽의 Y 축은 100을 기준으로 상대적인 높이입니다. 가장 큰 피크를 100으로 잡고 나머지는 상대적인 값으로 높이가 측정됩니다. 그러므로 주위에 어떤 높은 펩타이드가 존재하느냐에 따라 높이가 작은 펩타이드가 검출이 될수도 있고 검출이 안될수도 있습니다.만약 가장 높은 피크가 background 피크라면 아주 낮은 농도의 펩타이드들은 스펙트럼에서 보이지 않게 됩니다. 스펙트럼 상에서 보이지 않으면 DDA mode에서 fragmentation이 일어날수 없습니다. 이와 같은 원리로 같은 펩타이드들이지만 양이 상대적으로 많은 펩타이드가 존재할 경우 낮은 양의 펩타이드들이 또한 보이지 않게 됩니다. 이렇게 FAIMS는 LC에서 분리되는 펩타이드의 그룹을 몇개로 나누어 질량분석기로 들어가게 함으로써 농도가 아주 작은 펩타이드가 검출된 기회를 제공합니다.
FAIMS 에서 Method를 설정하는 방법
아래는 FAIMS를 장착하지 않았을때 사용하는 일반적인 DDA method입니다. 한개의 실험방법으로 진행합니다.

.하지만 FAIMS에서는 각각의CV별로 method를 만들어야 됩니다. 아래와 같이 experiment이 나누어져있습니다. 모든 파마미터는 동일하지만 FAIMS의 CV값만 다릅니다. 질량분석기에 들어오기전에 해당 CV값에 안정적인 펩타이드가 들어온후 그 다음은 동일한 방법으로 DDA 분석을 수행합니다. 단 CV에 따라 들어오는 펩타이드의 종류만 다르게 됩니다.

서로 다른 CV값을 사용하면 더 많은 펩타이드들의 종류가 따로 분리되어서 분석되겠지만 무작정 많은 CV 값을 사용할수 없습니다. Method 에서 보았듯이 CV값이 많아 질수록 그 만큼 한번의 cycle에 소요되는 시간이 증가됩니다. FAIMS가 없을때는 한번만 할 일을 FAIMS 를 사용하면 동일한 과정을 여러번 반복해야 됩니다. 그래서 FAIMS를 이용해서 여러번 scan을 하더라도 FAIMS가 없는 상태에서 한번만 하는것보다 좋은 결과를 가질 정도의 CV 수를 사용해야 합니다. 보통 2개 혹은 3가지의 CV 값을 사용합니다. 논문을 보면 서로 다른 CV 값을 사용하여 최적의 값을 찾는 실험이 많습니다. 제한된 cycle time에서 최대한 많은 CV 값 혹은 최적의 CV값을 찾기 위함입니다.
아래는 FAIMS로 얻어진 크로마토그램입니다. 기존의 크로마토그램과 다르게 보이지 않습니다.

하지만 이것은 3개의 CV값으로 얻어진 크로마토그램이 합쳐진것입니다.

FAIMS에서 펩타이드가 잘 분리가 되었다는것은 HPLC에서 retention time separation이 된것이 아니라 CV값에 따라 서로 다른 그룹에서 검출이 되었다는 것입니다.
아래는 이전 글에서 보여준 서로다른 CV값에서 얻어딘 N-linked glycopeptides 크로마토그램입니다. 크로마토그램에서 보듯이 CV값에 따라 intensity가 상당히 다릅니다. 피크가 보이지 않는것은 해당 CV값에서 Glycopeptide가 불안정하여 FAIMS를 통과하지 못하였기 때문입니다. LC에서는 용리가 되었으나 FAIMS에서 갇혀버렸습니다. 만약 주어진 시료에서 대부분의 non-glycopeptide들이 CV -40에서 안정적이고 N-linked glycopeptides가 CV -50에서 안정적이라면 N-linked glycopeptide들을 보다 잘 검출할수 있습니다.

Targeted Assay(PRM)
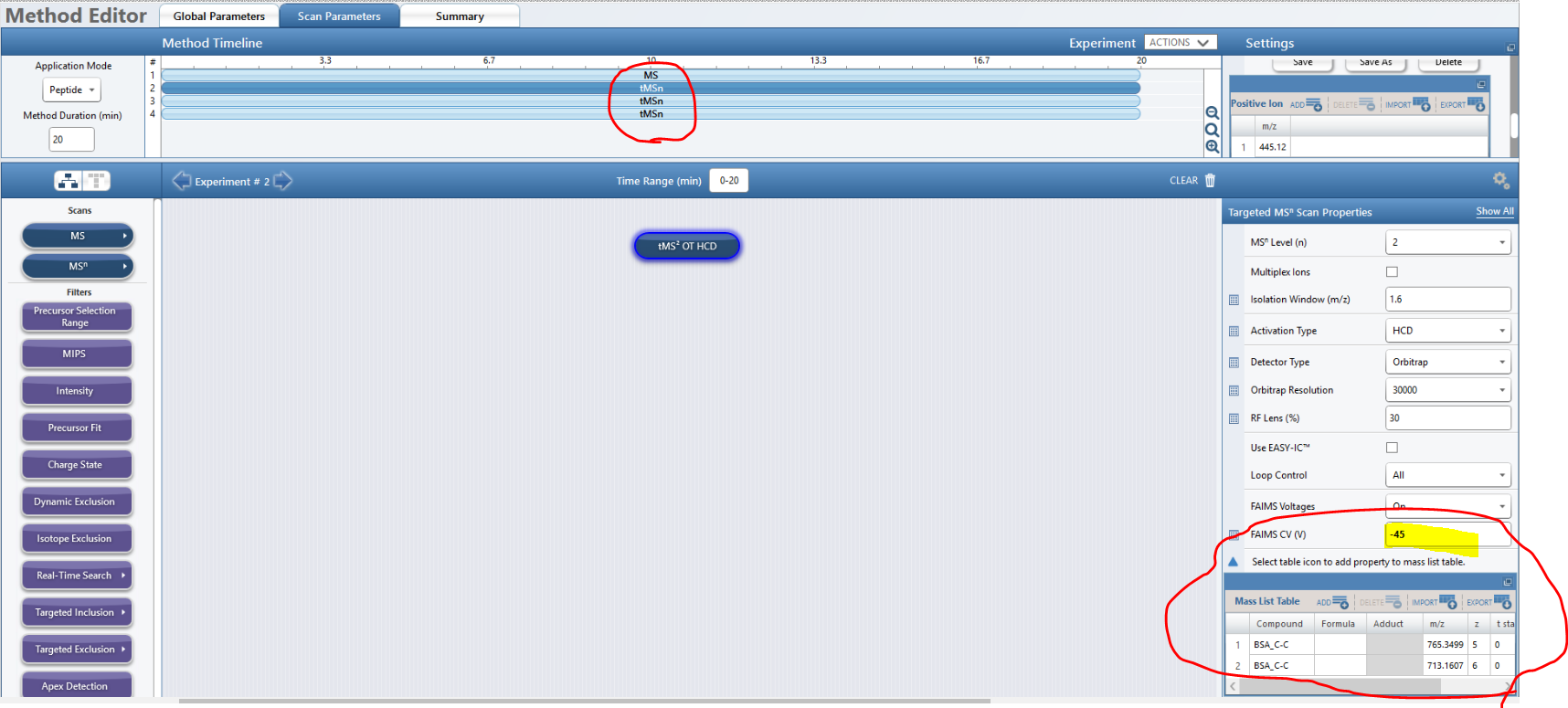
FAIMS를 이용하여 PRM을 수행하는것은 상당히 까다로울듯합니다. 일반적인 PRM 분석시 원하는 펩타이드 리스트는 한개의 method안에 삽입합니다. 하지만 FAIMS를 사용할때는 해당 펩타이들이 어느 CV 값에서 잘 나오는지 먼저 확인을 해야 합니다. 그리고 해당 CV값에 해당하는 PRM method를 개별로 만들어야 합니다. 해당 펩타이드의 수가 많고 상당히 다른 CV값에서 검출된다면 더 복잡하게 됩니다. 아래는 3가지의 CV값으로 PRM method를 만든 예입니다. CV값이 바뀔때마다 약간의 시간의 소요되기 때문에 많은 CV값을 하게 되면 아마 역효과가 날듯합니다.
관련글
Orbitrap Eclipse 와 FAIMS가 만나다
특별히 장비를 업그레이드를 하지 않고도 감도를 증가시킬수 방법이 있다면 좋을듯합니다. FAIMS도 그중에 하나가 아닐까 생각합니다. 새로운 기술을 위해 Bruker사의 Ion mobility 장비를 구매하
proteomicstechnology.tistory.com
FAIMS를 이용하여 선택적으로 N-linked glycopeptides 검출하는 방법
FAIMS를 이용하여 선택적으로 N-linked glycopeptides 검출하는 방법
펩타이드 분석에 있어서 FAIMS의 큰 장점중 하나는 CV값에 따라 선택적으로 펩타이드를 검출할수 있다는것입니다. 특히 서로 다른 Charge state를 가지는 펩타이드가 FAIMS에서 분리가 된다는 것입니
proteomicstechnology.tistory.com
FAIMS의 활용성
Orbitrap Eclipse에 FAIMS를 장착하기 전까지는 FAIMS의 활용성에 대해서 큰 확신이 없었습니다. 사실 Eclipse자체만으로도 대부분의 실험을 다 할수 있습니다. FAIMS가 없어서 하지 못하는 실험은 없을
proteomicstechnology.tistory.com
'프로테오믹스(단백체학)' 카테고리의 다른 글
| FAIMS를 이용한 DIA분석에서 고려해야 될것들 (0) | 2022.05.25 |
|---|---|
| FAIMS Inner/Outer electrode 세척 (1) | 2022.05.22 |
| Proteome Discoverer Workshop (June 14-17, 2022)/무료 온라인 (0) | 2022.05.20 |
| Sutter Instrument P-2000의 두가지 모델(P-2000/G and F) (0) | 2022.05.18 |
| Canonical vs Canonical & Isoform fasta file (0) | 2022.05.14 |




댓글