동일한 시료를 Data Dependent Acquisition mode로 여러분 분석시 처음에 검출된 펩타이드는 다음 분석에서 검출이 안되도록 설정하며 새로운 펩타이드가 검출된 가능성을 높일수 있습니다. 이때 사용하는것이 Exclusion mode입니다. 간단하게 앞에서 검출된 펩타이드의 m/z값과 retention time를 삽입하면 됩니다. C의 재현성에 따라 retention time window는 좌우 각각 1분 혹은 그 이상으로 설정하면 됩니다. 이것은 iontrap에서는 효과가 별로 없고 high resolution이 가능한 장비에서 유용합니다. 주어진 시간에서 m/z 값이 10 ppm(변경가능)이내에서 일치하는것만 exclusion 시키도록 설정합니다.
RT window를 넓게 하면 분석중에 RT가 shift됨에 따라 생길수 있는 오류를 방지 할수 있습니다. 하지만 너무 넓게 하면 오히려 해가 될 수 도 있습니다. Exclusion mode는 charge state를 구별하지 않습니다. 서로 다른 펩타이드임에도 불구하고 charge state는 다르나 monoisotopic m/z가 우연히 같은 펩타이드가 유사한 시간 내에 용리될수 있기 때문입니다(물론 가능성은 매우 낮습니다)
Proteome Discoverer에서 간단히 exclusion list를 생성할수 있습니다.
- Proteome discoverer에서 검색한 후 결과 파일을 엽니다.
- Peptidegroup에서 file-export-원하는 장비에 대한 mass list로 export 시킵니다.
- 원하는 펩타이드를 미리 선택하여 선택적으로 할수 있고 또한 전체 펩타이드를 모두 export 시킬수 있습니다.
- 원하는 RT winodw를 선택합니다.
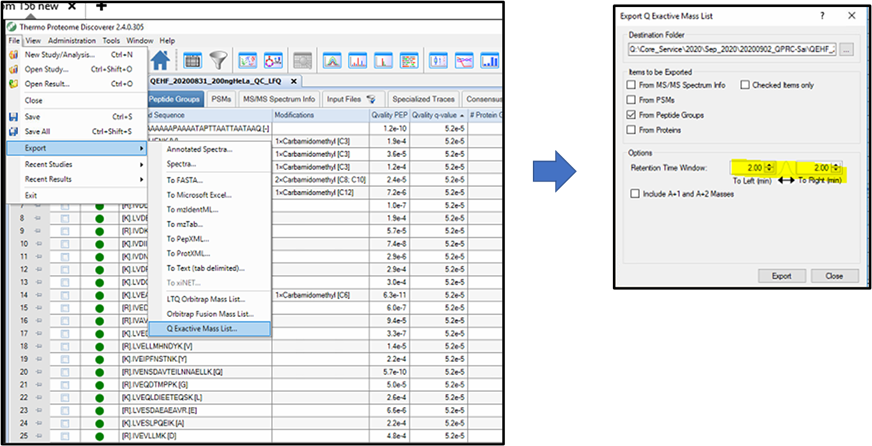
생성된 파일을 해당 장비의 method file에서 import 시킬수 있습니다. Q-Exactive로 예를 들어보겠습니다.
- 아래와 같이 Exclusion을 클릭후 앞에서 생성한 파일을 불러옵니다.

- 자동적으로 아래와 같이 list가 생성됩니다. OK를 누르고 method를 저장하면 완료됩니다.

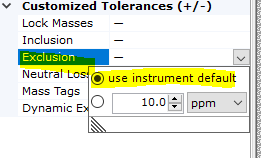
경우에 따라 list의 수가 제한이 있기 때문에 모두 첨가되지 않기도 합니다. Exclusion를 위한 tolerances를 장비 default 값을 따르게 되면 원하는 ppm을 설정해도 됩니다.
'프로테오믹스(단백체학)' 카테고리의 다른 글
| Biognosys 의 Spectronaut Pulsar 프로그램을 이용한 Spectra library 만들기 (0) | 2020.09.13 |
|---|---|
| ESI에서 Single Charged Peptides 검출을 위한 팁 (0) | 2020.09.10 |
| Short LC gradient에서 DDA 의 Top N 수 설정 (0) | 2020.09.06 |
| 많은 수의 human plasma 시료의 정량분석을 위한 전처리 방법 (0) | 2020.09.05 |
| Increasing the number of Plasma protein identification with a deglycosylation? (0) | 2020.09.03 |




댓글