반응형
얼마전에 Single charged가 많이 포함된 펩타이드 시료를 의뢰받았습니다. N-terminal 이 modification 이 되어있고 trypsin 이 아닌 다른 enzyme을 사용하여 digestion된 시료였습니다. 즉 N-term이 modification이 되어있고 C-term은 Basic 인 K 혹은 R 이 아닌 경우 ESI에서 single charge로 나올수가 있습니다.
ESI를 이용한 펩타이드 분석에서는 그 특성상 single charge 피크는 제외 시킵니다. background 나 chemical 피크들이기 때문이지요. 하지만 이 시료는 예외가 됩니다. 문제는 DDA 분석시 single charge 피크를 포함시킬 경우 많은 background 피크가 우선적으로 선택되어 실제 펩타드이가 많이 검출되지 않을수 있다는 것입니다.
(물론 Single charge 펩타이드가 background 피크보다 Intensity가 높다면 상대적으로 문제는 덜 할것입니다)
이와 같은 분석에서 약간의 Trick을 사용하여 분석하였습니다.
분석방법은 아래와 같습니다.
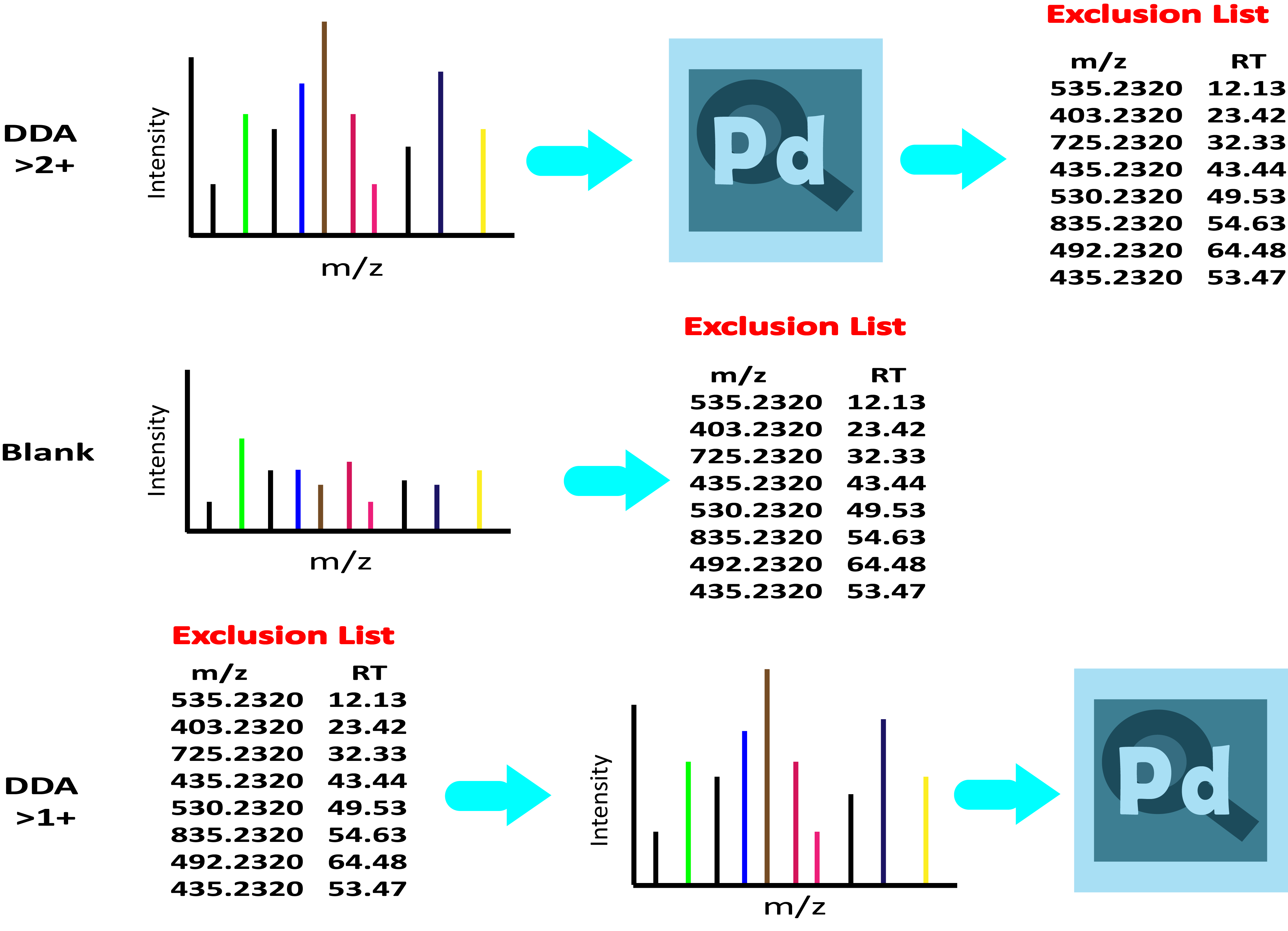
- 기존 방법대로 2+ 이상의 펩타이드를 먼저 분석합니다. ( >2+ DDA)
- Database 검색을 통해 검출된 펩타이드의 RT, m/z를 이용하여 exclusion list를 만듭니다. (Proteome Discoverer에서 Exclusion list를 만든는 방법)
- 대표적 background peaks 을 만들어진 exclusion list에 추가합니다. 여기에 두가지 방법이 있습니다. 기존에 알고 있는 대표적인 피크만 넣을수 있습니다. (e.g, m/z 445.1200 등). 또한 blank run을 수행하여 현재 장비에서 검출되는 background를 확인하여 리스트에 넣습니다. 물론 모든 background 피크를 다 넣기는 불가능함으로 Qualbroswer 에서 대표적으로 intensity가 높은 피크들을 선정하면 됩니다.
- 만들어진 2+ 이상의 검출된 펩타이드와 background 피크 리스트(m/z, RT)를 이용하여 최종 exclusion list를 만든후 method 에 삽입합니다.
- 이 method를 이용하여 동일한 시료를 한번 더 분석합니다. 이 방법을 이용하여 1+ 펩타이드의 검출 가능성을 높일수 있습니다.
이 방법을 이용하여 분석의뢰자의 시료에서 검출된 Single charge 펩타이드의 스펙트럼입니다 (아래)

'프로테오믹스(단백체학)' 카테고리의 다른 글
| Biognosys 사의 Spectronaunt Pulsar의 DirectDIA 분석 (without Spectra library) (0) | 2020.09.13 |
|---|---|
| Biognosys 의 Spectronaut Pulsar 프로그램을 이용한 Spectra library 만들기 (0) | 2020.09.13 |
| Proteome Discoverer에서 Exclusion list 만드는 방법 (0) | 2020.09.10 |
| Short LC gradient에서 DDA 의 Top N 수 설정 (0) | 2020.09.06 |
| 많은 수의 human plasma 시료의 정량분석을 위한 전처리 방법 (0) | 2020.09.05 |




댓글