
MS2-MS3 를 이용한 분석
Orbitrap Fusion에서의 Method는 아래와 같습니다. 요약하자면 CID fragmentation을 이용하여 전형적인 DSSO 피크를 찾습니다. 두개의 피크가 31.9721 Da 차이가 나는 MS2 스펙트럼이 있을 경우 각 두개의 피크에 대해서 MS3를 수행합니다.
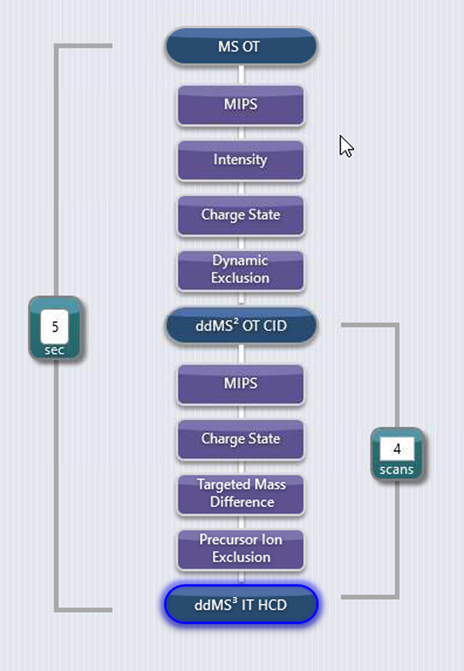
DSSO crosslinking 펩타이드만을 최대한 검출하기 위해서는 MS1에서 피크를 선택할때 2+는 제외하고 3-6로 설정하기도 합니다. 보통 crosslinking 된 펩타이드의 경우 높은 전하를 가집니다. 즉 일반적인 2+ 펩타이드에 의해서 검출기회를 놓칠수 있기 때문에 2+ 펩타이드는 제외시키는것이 좋습니다.

ddMS2 OT CID는 아래와 같습니다. 정확히 31.9721 Da (10ppm 이내) 차이나는 펩타이드를 찾아야 함으로 detection type은 Orbitrap를 선택해야 합니다.

Targeted Mass difference 에서는 아래와 같이 설정을 합니다. 31.9721 Da 차이가 나야하며 차이나는 두개 모두를 MS3로 수행하게 설정합니다 (both ions in the pair). 두개의 paired 펩타이드는 동일한 전하를 가져야 합니다 (Ion must be the same charge)

MS3는 아래와 같이 설정합니다. Activation type은 CID 혹은 HCD로 설정할수 있습니다. 그리고 detector의 경우 ion trap이 더 좋습니다. MS2를 수행하고 MS3를 수행하는 과정에서 이온들이 충분히 많지 않게 됩니다. 이 경우 ion trap으로 검출하는것이 감도면에서 더 좋습니다.

'프로테오믹스(단백체학)' 카테고리의 다른 글
| Orbitrap Fusion의 MS2-MS3를 이용한 DSSO crosslinking 펩타이드 검출방법 (0) | 2021.03.25 |
|---|---|
| DSSO crosslinking 펩타이드 분석을 위한 Proteome Discoverer- XlinkX 검색 설정방법 (0) | 2021.03.25 |
| TMT 정량분석에서 Proteome Discoverer의 Co-isolation threshold 값의 효과 (0) | 2021.03.22 |
| 혈액단백질 (Plasma & Serum)의 프로테오믹스 분석의 어려운점 (0) | 2021.03.18 |
| DSSO (disuccinimidyl sulfoxide) Crosslinking (XL-MS) 펩타이드 분석 (BSA+Cytochrome C) (0) | 2021.03.17 |




댓글