DSSO Crosslinking 펩타이드를 Orbitrap Fusion에서 MS3 방법으로 얻은 데이타를 Prteome Discoverer 에서 검색하기 위한 workflow입니다.
Process Workflow
Process workflow의 전체적인 절차는 아래와 같습니다.
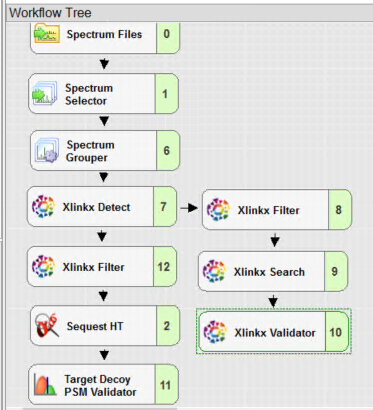
DSSO로 반응시킨 시료에는 DSSO가 결합된 펩타이드와 함께 결합되지 않은 일반 펩타이드가 존재합니다. 그래서 검색시 이 두 가지 펩타이드를 모두 검출하는 workflow가 필요합니다. 앞 단계는 동일하지만 XlinkX detector에서 Use MS(n-1) with Parent Precursor로 설정합니다 Crosslink Modification은 DSS/+158.005 Da를 설정합니다.
Xlinxk Filter 에서는 crosslinks와 Peptides를 설정할수 있습니다.


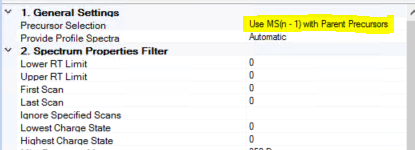
Peptides를 설정한 workflow에서는 바로 Sequest로 검색합니다. Dynamic modification에 3가지의 서로 다른 DSSO modification form이 있습니다. 아마 이것은 quenching시 무엇을 사용하느냐에 따라 다르게 설정하는것 같습니다.
이 부분은 정확히 몰라서 3가지 모두 입력하였습니다.
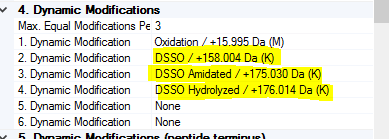
Crosslinks로 설정했을 경우 Xlinkx Search 를 통해 검색합니다. 아래는 설정 파라미터인데 Sequest에서와 유사합니다.
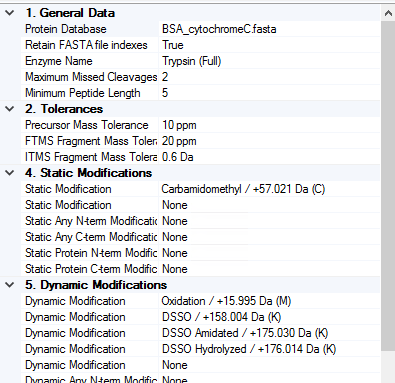
그리고 Xlinkx validation을 수행합니다. 아래는 default 값입니다.

Consensus Workflow
Consensus Workflow를 아래와 같습니다.

다른 workflow와 특별히 다르지 않고 Xlinkx Crosslink Grouping이 포함됩니다. 아래는 default 값입니다.
일단 이렇게 간략히 이렇게 시작해서 실험목적에 따라 최적화 하면 될것같습니다.
관련글
DSSO crosslinking 펩타이드 분석을 위한 Proteome Discoverer- XlinkX 검색 설정방법
DSSO (disuccinimidyl sulfoxide) Crosslinking (XL-MS) 펩타이드 분석 (BSA+Cytochrome C)
'프로테오믹스(단백체학)' 카테고리의 다른 글
| 150 µm ID (내경) 컬럼의 nano column으로 낮은 압력으로 펩타이드 분리하기 (0) | 2021.03.27 |
|---|---|
| Orbitrap Fusion의 MS2-MS3를 이용한 DSSO crosslinking 펩타이드 검출방법 (0) | 2021.03.25 |
| DSSO (disuccinimidyl sulfoxide) Crosslinking 펩타이드 분석을 위한 Orbitrap Fusion MS3 Method 설정 (0) | 2021.03.23 |
| TMT 정량분석에서 Proteome Discoverer의 Co-isolation threshold 값의 효과 (0) | 2021.03.22 |
| 혈액단백질 (Plasma & Serum)의 프로테오믹스 분석의 어려운점 (0) | 2021.03.18 |




댓글