이번에는 DSSO를 이용하여 crosslinking된 시료를 Orbitrap Fusion 의 MS2-MS3방식을 이용하여 검출한뒤 Proteome Discoverer로 검색된 결과를 살펴보겠습니다. 앞에서 Fusion MS2-MS3 방법과 Proteome Discoverer 검색 방법을 설명하였습니다.
관련글
DSSO crosslinking 펩타이드 분석을 위한 Proteome Discoverer- XlinkX 검색 설정방법
DSSO (disuccinimidyl sulfoxide) Crosslinking (XL-MS) 펩타이드 분석 (BSA+Cytochrome C)
Peptide Identification
먼저 Xlinxk Filter에서 Peptide로 설정된 부분에서 검춝된 펩타이드의 리스트를 보여주입니다. DSSO와 연결된 두개의 펩타이드가 아닌 일반 펩타이드와 DSSO가 PTM과 같이 붙은 형태의 펩타이드에 대한 정보를 보여줍니다.

예를 들면 아래 DSSO 가 Hydrolyzed 형태로 AEFVEVTKLVTDLTK 에 결합된 MS2 스펙트럼입니다.

DSSO crosslinking Peptide Identification
아래와 같이 DSSO로 결합된 두개의 펩타이드에 대한 정보 (예를 들면 Sequence, m/)등이 나옵니다.

아래와 같이 각각의 펩타이드에 대한 Sequence가 보입니다. 여기서는 하나의 BSA에 있는 서로 다른 펩타이드이 결합되었습니다.

아래는 BSA에서 두 개의 서로 다른 펩타이드가 DSSO로 연결된 Precursor ion의 피크를 보여줍니다. 높은 전하 (5+, m/z 754.9574)를 가지고 있습니다.
VHKEC(Carbamidomethyl)C(Carbamidomethyl)HGDLLEC(Carbamidomethyl)ADDRADLAK
-->MH+: 2612.1650, M: 2611.157 Da
ALKAWSVAR
--> MH+ : 1001.5891, M: 1000.5811 Da
(2611.157+1000.5811+158.0038+5)/5 =754.9574 (5+)
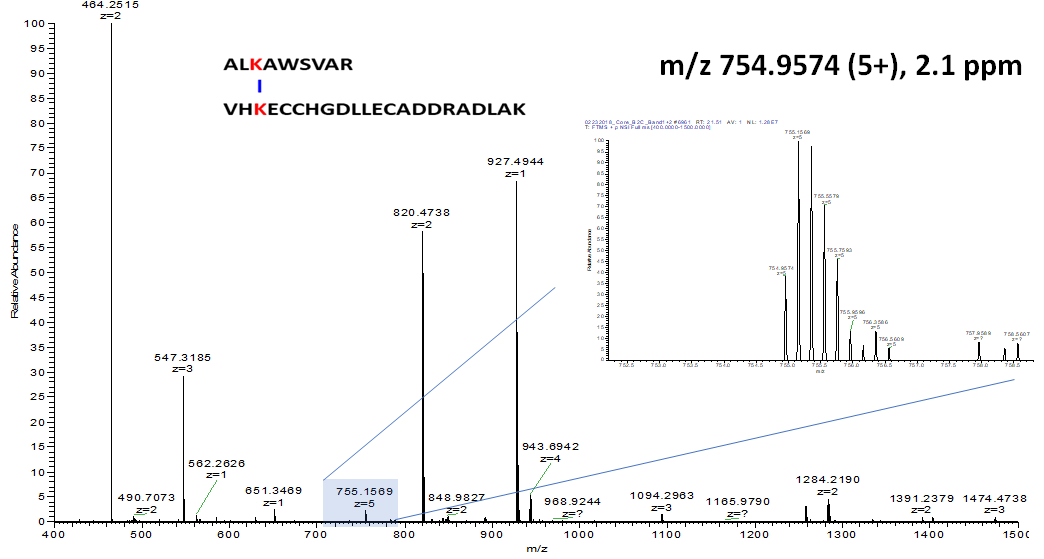
이 펩타이드에 대해서 DSSO의 전형적인 CID MS2 스펙트럼을 보여줍니다. DSSO로 결합된 두개의 펩타이드가 CID에서 절단되면서 생성된 전형적인 피크가 보입니다. 아래와 같이 두개의 전형적인 피크인 m/z 675.5429 (z=4) 와 m/z 667.550(z=4)가 보입니다. z=4일때 7.9924 Da 차이가 납니다. 즉 분자량은 31.9699 Da가 차이가 나며 이론적인 값(31.9721) 과 거의 일치합니다.

Orbitrap fusion의 method에서 이 펩타이드는 선택적으로 MS3를 수행하며 펩타이드를 검출하게 됩니다. 아래 스펙트럼은 실제 raw 파일에서의 스펙트럼입니다.

위의 두개의 피크에 대해 각각 MS3를 수행였습니다. 아래는 raw 파일에서 얻어진 MS3 스펙트럼입니다.


즉 이렇게 MS2와 MS3가 수행되어서 얻어진 결과를 토대로 Proteome discoverer가 검색하여 결과를 보여주게 됩니다.
단일 단백질에서는 상대적으로 검출이 가능할수 있을것 같습니다. 하지만 여러 단백질이 혼합된 시료의 경우 Proteome discoverer가 제대로 검출을 할수 있을지는 모르겠습니다. 아마 혼합시료의 경우 SCX, SEC등을 통해서 DSSO crossliking펩타이드만을 선택적으로 회수하는것이 필요할것입니다.
'프로테오믹스(단백체학)' 카테고리의 다른 글
| Radio frequency (RF) Lens(%) 값에 따른 서로 다른 m/z 이온들의 감도 변화 (0) | 2021.03.28 |
|---|---|
| 150 µm ID (내경) 컬럼의 nano column으로 낮은 압력으로 펩타이드 분리하기 (0) | 2021.03.27 |
| DSSO crosslinking 펩타이드 분석을 위한 Proteome Discoverer- XlinkX 검색 설정방법 (0) | 2021.03.25 |
| DSSO (disuccinimidyl sulfoxide) Crosslinking 펩타이드 분석을 위한 Orbitrap Fusion MS3 Method 설정 (0) | 2021.03.23 |
| TMT 정량분석에서 Proteome Discoverer의 Co-isolation threshold 값의 효과 (0) | 2021.03.22 |




댓글