종종 Disulfide-linked Peptides 분석에 대한 의뢰가 들어옵니다.. 하지만 disulfide bond 펩타이드 profiling 분석은 일반 분석서비스로 진행하기가 아직은 어렵습니다. 기존의 Database program으로 자동검색이 거의 불가능합니다. 또한 ETD와 같은 특별한 Fragmentation 모드가 필요하지만 만 최적화하기도 쉽지 않습니다.
만약 이미 예상한 Disulfide-linked Peptides 가 있을 경우에는 이를 확인하는 차원에서는 가능합니다. 왜나하면
각각의 펩타이드의 m/z 값을 알고 있으면 수작업으로라도 확인할수 있기 때문입니다. 아무튼 차후 분석의 최적화를 위해서 간단히 테스트를 해보았습니다. 기존 논문의 BSA를 이용한 연구결과를 참고 하였습니다.
먼저 BSA power를 reduction/ alkylation 과정없이 바로 trypsin digestion 하였고 비교 분석을 위해 기존 방법대로 reduction/alkylation 후 tryspin digestion 처리하였습니다. 아래는 Reduction/Alkylation 한것과 하지 않고 trypsin digestion한 BSA의 base-peak chromatogram입니다.

크로마토그램에서 보듯이 두 피크들의 패턴이 상당히 다름을 알 수 있습니다.. Disulfide-bond를 유지하고 있을 경우 펩타이드에 해당하는 피크의 수가 줄어 들었습니다. 또한 스펙트럼을 살펴보면 reduction/alkylation 에서는 보이지 않는 높은 전하를 가지는 피크들이 아래와 같이 많이 보입니다.. (Figure 2). 아마 이러한 펩타이드가 disulfide-linked 형태임을 예상할수 있습니다.

앞에서도 언급했듯이 이러한 피크들에 대한 MS/MS 스펙트럼이 존재하더라도 분석할수있는 적절한 프로그램이 없습니다. 논문에 나와있는 프로그램들은 실제로 사용하면 잘 안되는 경우가 많고 아주 좋은 스펙트럼에서만 적용되는 경우가 대부분입니다.
다행히 BSA 경우 기존의 논문에 분석된 데이타가 많음으로 그 데이타를 이용하여 manual assignment해보기로 하였습니다. 기존의 논문에 있는 정보와 일치하는 m/z 와 charge state를 가지는 펩타이드를 선정한후 targeted assay (PRM)로 high-quality MS/MS 스펙트럼을 얻었습니다(아래그림 참조). High-quality MS/MS 스펙트럼은 manual assignment하는데 유용합니다.

기존의 논문에서 대표적인 두개의 펩타이드를 정한후 PRM 분석으로 얻어진 MS/MS 스펙트럼을 분석하였습니다.
아래 그림은 manual assignment를 통해 얻어진 Intra-, Inter-disulfide-linked peptides 스펙트럼입니다. 그림에서 보듯이 수작업을 통해 disulfide bond 펩타이드를 확인할수 있습니다.

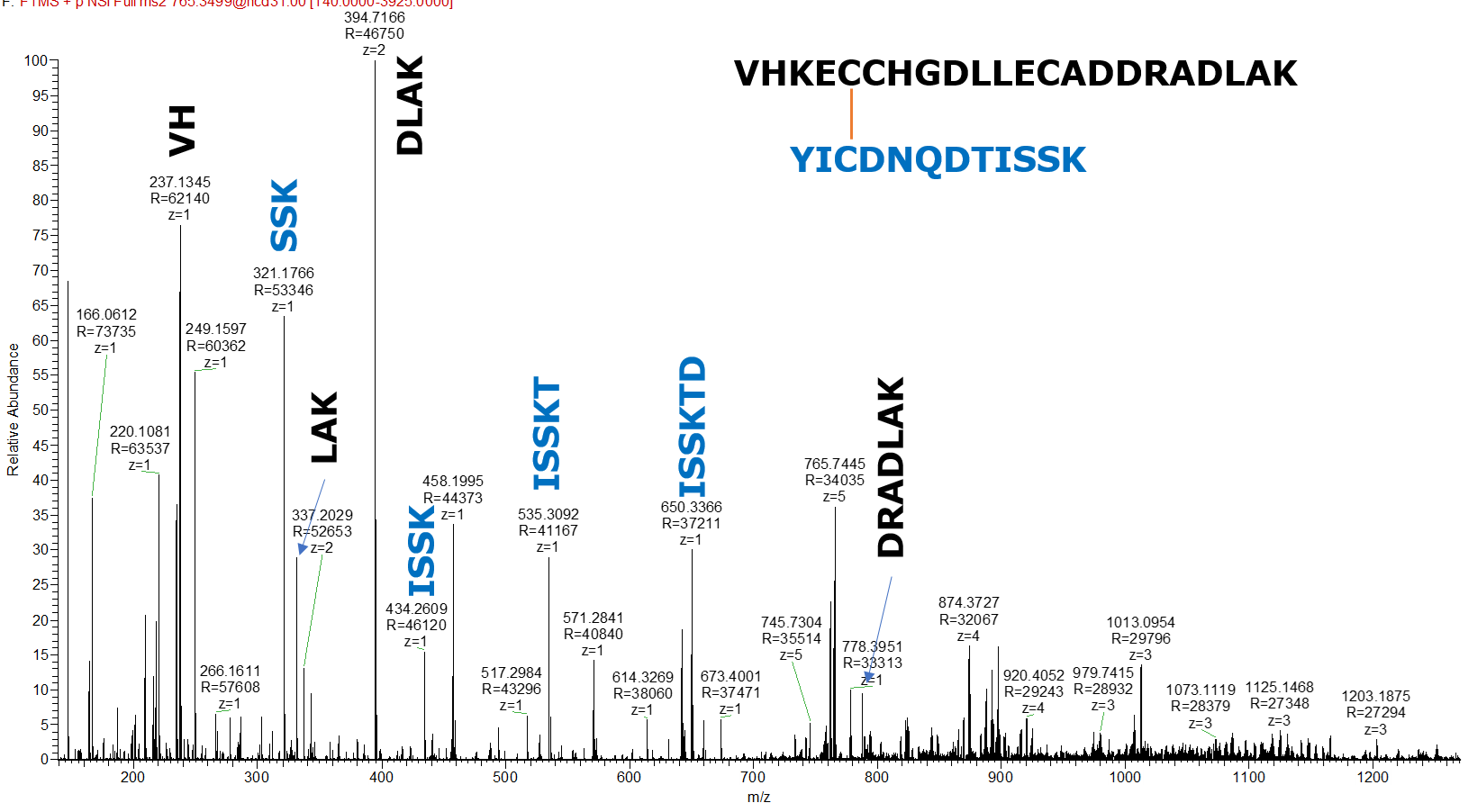
사전에 알고있거나 예상되는 Disulfide-linked 펩타이의 경우 이러한 접근법으로 실제 존재여부 및 구조분석이 가능할것 같습니다.
'프로테오믹스(단백체학)' 카테고리의 다른 글
| S-Trap™ - ProtiFi 를 이용한 단백질 정제 효율성 검증 (2) | 2020.04.18 |
|---|---|
| Easy nLC and Dionex LC 의 Valve Stator의 Scratch 현상 확인하는 법 (0) | 2020.04.17 |
| 단일 In-gel 밴드에서 99% Sequence coverage 얻기 (0) | 2020.04.16 |
| Promega 6 × 5 LC-MS/MS Peptide Reference Mix 를 이용한 시스템 QC (0) | 2020.04.15 |
| Active Background Ion Reduction Device (ABIRD)를 이용한 Background Peaks의 감소 측정 (0) | 2020.04.14 |




댓글