Immunoprecipitation (IP)를 한 시료의 경우 예상되는 단백질이 존재합니다. 분석의뢰자는 당연히 그 단백질만 검출될것이라고 예상합니다. 간혹 아주 높은 Sequence coverage로 검출되기를 기대합니다.
특정 단백질이 예상될 경우 데이타 분석시 간편함을 위해서 또는 sequence coverage를 높이기 위해서 해당 단백질만의 Sequence만으로 검색을 합니다. 하지만 이 경우 Sequence coverage는 높아질 가능성이 있지만 false-positive가 더 많이 생성될수 있습니다.
비록 IP를 수행하더라도 완전히 순수한 단백질만을 회수하기는 어렵습니다. 또한 human 시료의 경우 Keratin 불순물로 같이 검출될 가능성이 있습니다. 작은 DB를 사용할 경우 분석오류가 발생할수 있으며 또한 해당 펩타이드 피크가 아님에도 불구하고 한개의 DB에서 찾는 과정에서 강제적으로 matching 시키는 경우도 있습니다. 즉 이러한 가능성을 배제하고 오로지 한가지 DB를 사용하게 되면 문제점이 발생할수 있습니다.
최근에 분석한 IP를 수행하여 Single SDS-PAGE 밴드로 제공한 시료를 분석하였습니다.
먼저 해당 단백질 fasta 파일만으로 검색하여 총 66개의 unique 펩타이드가 검출되어 92% sequence coverage를 얻었습니다.

동일한 raw 파일을 Human fasta 파일로 검색을 하였습니다. 아래 보는것과 같이 해당 단백질외에 다른 단백질, 특히 Keratin 단백질이 많이 검출되었습니다. 해당 단백질은 45개의 unique 펩타이드가 검출되어 79% sequence coverage를 얻었습니다.

동일한 raw 파일임에도 fasta 파일의 크기에 따라 다른 결과를 보입니다. human fasta 파일로 검색하였을때 검출되지 않았지만 해당 단백질 fasta파일에서 검출된 펩타이드의 MS/MS 스펙트럼을 확인해보았습니다.
이 펩타이드는 약 30개 아미노산으로 구성된 분자량이 약 3300 Da의 펩타이드 입니다. 하지만 Matching 된 MS/MS 스펙트럼은 아주 좋지 않습니다. 몇개의 fragments만 검출되었을 뿐만 아니라 matching 된 피크도 major peak가 아닌 거의 background 피크들에 해당됩니다. 검출된 retention time 은 55분 경입니다. 실제 60분의 gradient 중에서 펩타이드가 다 용리되고 washing 되는 영역입니다. 이 영역에서 많은 지저분한 펩타이드가 아닌 불순물 피크가 많이 나옵니다. 이러한 피크들중에서 오류로 인해 검색이 되는 경우가 많습니다.
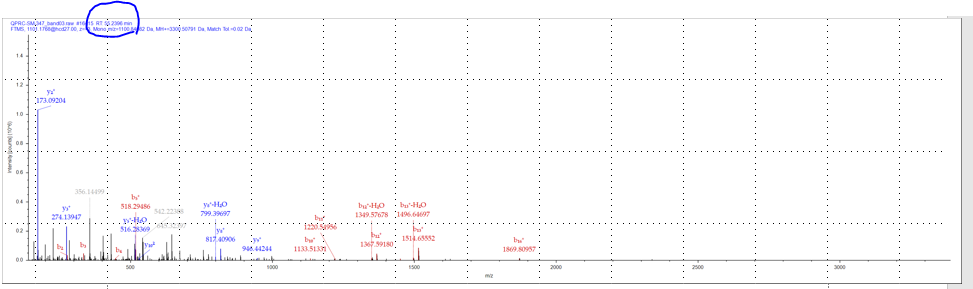
분석 의뢰자들에게 높은 sequence coverage를 제공할 목적으로 single fasta 파일을 사용할 경우가 종종 있습니다. 하지만 위의 설명한 데로 문제가 발생할 경우가 많기 때문에 특별한 경우가 아니면 추천하지 않습니다.
아래는 human cell lysate를 profiling 한후 human database로 검색하여 검출한 단백질중 하나입니다. 아래는 100% Sequence coverge를 얻었습니다.

'프로테오믹스(단백체학)' 카테고리의 다른 글
| Herceptin, monoclonal Antibody 의 Disulfide bond 펩타이드 분석법 (2) | 2020.12.21 |
|---|---|
| Monoclonal Antibody (mAb)에서 Intact N-linked glycopeptides 분석 (0) | 2020.12.17 |
| HPLC 이동상 (buffers)의 오염으로 인한 background peaks (0) | 2020.12.03 |
| Histone Propinolylation을 통한 bottom-up proteomic analysis (0) | 2020.12.01 |
| PRM / DIA 정량분석법의 최적화를 위한 peak point 찾기 (실제 데이타) (0) | 2020.11.18 |




댓글