정성 분석을 위한 DDA 실험에서는 주어진 시료에서 최대한 많은 펩타이드를 검출하는것이 목적입니다. 이러한 이유로 펩타이드 피크 당 1~2번 정도의 MS/MS 스펙트럼만 있으면 됩니다. 더 많다고 더 좋은것은 아니며 오히려 전체 검출단백질수는 줄어들게 됩니다. 이론적으로는 아주 좋은 MS/MS 스펙트럼 한개만 있는것이 좋습니다. 하지만 정량분석은 성격이 다릅니다 정량분석을 위해서는 특정 시점의 피크 높이를 측정하는것이 아니라 펩타이드가 용리되는 시간동안의 피크의 면적을 측정해야 됩니다. 이에 대한 설명은 앞의 글에서 적었습니다. 이번에는 간단한 실험을 통해서 분석방법에 따라 peak point수가 어떻게 달라지는지 보겠습니다. 100fmol BSA를 Q-Exactive로 분석하였습니다.
Target peptide 수에 따른 point 수
동일한 PRM 조건에서 target 펩타이드의 수를 변화시켜 측정해보았습니다. 동일한 질량분석기의 파라미터를 가진다면 target 펩타이드의 수가 많아 질수록 한 cycle를 왕복하는데 시간이 더 많이 걸리게 됩니다. 아래 크로마토그램은 동일한 PRM 조건에서 각각 5, 10, 15, 20, 50 개의 BSA target 펩타이드를 분석한것입니다.

자세하게 보기 위해서 5개와 50개 펩타이드를 분석한 PRM에 대한 크로마토그램을 보겠습니다. 5개 펩타이드를 분석한것이 50개를 분석한것에 비해서 훨씬 촘촘한 피크형태를 보여줍니다.

5 펩타이드 분석의 경우 Full MS 후 5번의 MS/MS를 수행한후 다시 Full MS를 진행합니다. 반면에 50개의 펩타이드의 경우 한개의 Full MS 수행후 50번의 MS/MS를 수행하도록 설정하였기 때문에 훨씬 덜 촘촘하게 보입니다. 왜나하면 MS/MS의 TIC는 상대적으로 Full MS의 TIC에 비해서 훨씬 낮기 때문입니다.

이제 특정 펩타이드의 피크에 대한 point수를 확인해보겠습니다. 아래 그림은 5가지의 분석방법에 대해서 대표적인 펩타이드 대한 XIC를 보여줍니다. Xcalibur Qualbrower에서 마우스를 움직이면서 대략의 peak point 수를 세어보았습니다. 피크 오른쪽에 tailing이 있기 때문에 일정구간까지만 포함시켰습니다. 그림에서 보이듯이 5, 10, 15, 20, 50개로 target 펩타이드수가 증가할 수록 point수는 조금씩 줄어들었습니다.

cycle time은 질량분석기의 속도, maximum injection time, LC peak 간격등을 이용하여 peak point를 대략 계산해 낼수 있습니다. 하지만 위에서 본것 처럼 역으로 raw 파일을 이용하여 수작업으로 대략 peak point 수를 확인할수 있습니다. 이런 방식을 이용하면 적절한 peak point수를 확보하기 위해서 target peptide 수를 증가시키거나 줄일수도 있습니다.
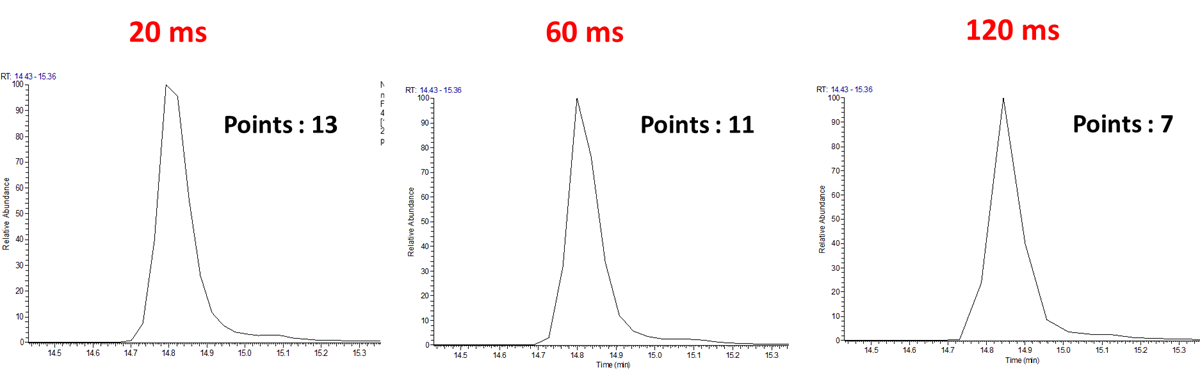
Maximum injection time의 변화에 따른 peak point 수 측정
다음은 동일한 target peptide수와 AGC값을 이용하여 서로 다른 MS/MS maximum injection time (MIT) 에 따른 peak point수를 비교해 보았습니다. 특정 이온이 설정한 이온 수 만큼 채워지지 않을 경우 무한정 기다리는것이 아니라 MIT 시간 만큼 최대한 기다린후 다음으로 넘어갑니다. 그래서 펩타이드의 양에 따라 MIT 시간을 다 사용할수도 있고 그렇지 않을수 도 있습니다. 그래서 정확한 비교는 되지 않겠지만 아래와 같이 그림에서와 같이 20 target 펩타이드에서 20, 60, 120 ms 의 MIT를 사용했을때 peak point수는 13, 11, 7로 조금싹 감소하였습니다. 설정한 MIT 값의 차이에 비해서는 point수가 확연히 차이가 나는건 아닌것 같습니다.
LC peak resolution에 따른 peak point 수
이번에는 모든 조건이 동일할때 peak의 넓이에 따른 point의 수를 비교해보았습니다. 이를 위해서 동일한 조건에서 서로 다른 LC gradient (15, 30, 60min)를 사용하였습니다. LC gradient가 증가함에 따라 피크가 넓어지는 효과가 있기 때문입니다. 아래는 LC gradient에 따른 peak point수를 보여줍니다. LC gradient의 시간이 증가함에 따라 peak의 넓이가 넓어지는것을 볼수 있고 이로 인해 peak point 수는 증가되는것을 볼수 있습니다.

하지만 peak의 intensity는 3E7, 2E7, 8E6로 각각 감소되었습니다. 그러므로 peak point수가 증가되는것만이 좋은것이 아닙니다. 먼저 최적의 LC 조건을 확립한후 peak 모양에 따라 적절한 peak point 수를 가질수 있는 조건을 찾아야 합니다. 약간 번거롭지만 이렇게 raw 파일만으로 자신의 PRM (혹은 DIA) 방법이 괜찮은것인지 확인 할 수 있습니다.
관련글]DIA분석(QE)에 있어서 m/z 500-900 과 mz 400-1000 검색구간에 따른 결과 차이
관련글] Skyline의 DIA method 설정에서 Window 수와 Optimize window placement 에 따른 비교 분석 (QE-HF)
관련글] 적은수의 펩타이드를 정량분석에 있어서 PRM과 DIA 분석법 비교
관련글] PRM / DIA 정량분석법의 최적화를 위한 peak point 찾기 (실제 데이타)
관련글] PRM / DIA 정량분석법의 최적화를 위한 peak point 찾기
관련글] Biognosys Spectonaut directDIA vs Gas-phase Fractionationed based EncyclopeDIA
관련글] Spectronaut의 direct DIA 분석 검증(Human plasma &Achilles tendon)
관련글] Spectronanut의 directDIA 분석과 Spectra library 기반의 정량분석 비교
관련글] Biognosys Spectronaut 의 Pulsa, directDIA를 이용한 DIA 검색능력 비교
관련글] Biognosys 사의 Spectronaut Pulsar의 DirectDIA 분석 (without Spectra library)
관련글] Biognosys 의 Spectronanut Pulsar 프로그램을 이용한 Spectra library 만들기
관련글] DIA Spectra library를 위한 Spin-column 형태의 hpRP, SCX, and HILIC 기반 펩타이드 분획
관련글] EncyclopeDIA, Spectronaut, PD2.4 with Overlap & non-Overlap DIA and DDA analysis
관련글] Overlap DIA 파일을 HTRMSConverter 로 변환하여 Spectronaut 분석하는 방법
관련글] EncyclopeDIA 을 이용한 DIA 단백질 정량분석
관련글] [Biognosys spectronaut] DIA 분석에서 Spectra library에 따른 단백질 검출차이
관련글] [Biognosys spectronaut] Configure condition 설정
관련글] Spectronaut 의 directDIA 분석과 Spectra library 기반의 정량분석 비교
관련글] Spectronaut 의 direct DIA 분석 검증(Human plasma &Achilles tendon)
관련글] [Biognosys spectronaut] DIA normalization Setting (Ovalbumin DIA 정량분석)
관련글] EncyclopeDIA, Spectronaut, PD2.4 with Overlap & non-Overlap DIA and DDA analysisEncyclopeDIA, Spectronaut, PD2.4 with Overlap & non-Overlap DIA and DDA analysis
'프로테오믹스(단백체학)' 카테고리의 다른 글
| HPLC 이동상 (buffers)의 오염으로 인한 background peaks (0) | 2020.12.03 |
|---|---|
| Histone Propinolylation을 통한 bottom-up proteomic analysis (0) | 2020.12.01 |
| PRM / DIA 정량분석법의 최적화를 위한 peak point 찾기 (0) | 2020.11.18 |
| LC-MS/M에서 감도를 높이기 위해서는 LC 분리능이 좋아야 하는 이유 (0) | 2020.11.11 |
| 나의 손을 거처간 질량분석기들 이야기 (0) | 2020.11.07 |




댓글