요즘은 다시 DIA를 기초부터 공부하는기분입니다. 이론적으로 알고 있거나 논문에 이미 결과가 나와 있더라도 직접 운용하는 장비에서는 어떠한지 확인이 필요하다고 생각합니다. 그리고 결과를 정리해 놓아야 다음에 다른 분석에 적절히 사용할수 있을 것 같습니다.
초기에는 DIA의 분석구간을 m/z 500-900으로 하였습니다. 일반적으로 DDA에서 사용하는 m/z 300-1500 혹은 m/z 400-1200을 사용하지 않은것은 초기 장비의 한계로 인해 최대한 검색 구간을 줄여서 감도를 향상시키기 위함이 었을 것입니다. 많은 논문에서 Q-Exactive series의 초기 모델인 QE의 경우 이렇게 m/z 500-900 구간을 사용했던 것 같습니다. 보통 tryptic peptide의 경우 m/z 500-1000 사이에서 많이 검출되며 그 외 구간에서는 상대적으로 적게 검출됩니다. 그러므로 적은 수의 펩타이드를 더 검출하기 위해 감도를 희생하지 않는것이 좋다고 생각합니다.
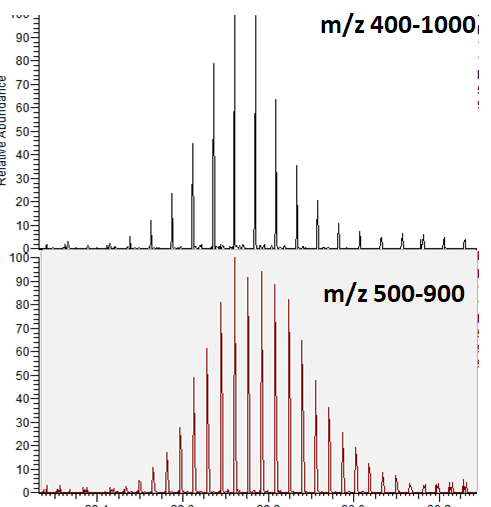
아래 그래프는 293T cell을 stage-tip으로 분획하여 QE로 검출후 피크와 펩타이드 m/z의 분포도를 보여줍니다.
검출된 피크와 실제 펩타이드로 검색이 된 피크의 분포도에서 보듯이 m/z 1000 이상에서는 매우 적은양이 검출되었습니다. m/z 400-500 구간은 상대적으로 많이 검출이 되었습니다.

실험실에 QE가 있어서 간단히 m/z 500-900 와 m/z 400-1000 두 구간에서의 결과를 비교해보았습니다.
2 µg 293T cell digest를 20 m/z isolation window로 두개의 다른 구간에서 3번 반복 분석하여 결과를 얻었습니다
아래의 그래프에서 보듯이 검출된 단백질의 숫자에는 큰 차이가 없지만 넓은 구간(m/z 400-1000)에서 더 많은 펩타이드가 검출되었습니다. 더 넓은 구간에서 분석하였으니 더 많은 펩타이드가 검출된것은 당연한 결과입니다. 하지만 검출된 펩타이드가 새롭게 검출된 단백질의 갯수를 증가시키지는 못하였습니다. 아마 다른 charge state 를 가지는 동일한 펩타이드가 검출된것이라 생각합니다.

시료에 spiking을 시킨 Ovalbumin 펩타이드의 검출결과입니다. 테이블에서 보듯이 넓은 구간에서 검출된 펩타이드의 수가 더 많습니다. 하지만 동일한 펩타이드의 다른 charge state를 가지는것도 있습니다. 예를 들면
ADHPFLFC[Carbamidomethyl]IK 의 경우 3+는 m/z 416.5462 입니다. 즉 이 펩타이드는 m/z 500-900 구간에서는 검출이 되지 않고 m/z 400-1000 구간에서만 검출이 됩니다.

일단 검출된 수만 비교하면 m/z400-1000에서의 결과가 더 좋습니다.
아래는 특정 product ion의 XIC를 보여집니다. 그림에서 보듯이 m/z500-900영역에서는 더 많은 포인트수로 검출이 되었습니다. 즉 구간이 줄어들면 그 만큼 해당 피크의 검출 횟수가 증가됩니다. 정량분석에서는 이 점이 중요합니다.
하지만 m/z 400-1000에서도 대략 10개 이상의 포인트로 검출이 되었으로 정량분석의 정확도에는 큰 문제가 없을것 같습니다.
관련글]DIA분석(QE)에 있어서 m/z 500-900 과 mz 400-1000 검색구간에 따른 결과 차이
관련글] Skyline의 DIA method 설정에서 Window 수와 Optimize window placement 에 따른 비교 분석 (QE-HF)
관련글] 적은수의 펩타이드를 정량분석에 있어서 PRM과 DIA 분석법 비교
관련글] PRM / DIA 정량분석법의 최적화를 위한 peak point 찾기 (실제 데이타)
관련글] PRM / DIA 정량분석법의 최적화를 위한 peak point 찾기
관련글] Biognosys Spectonaut directDIA vs Gas-phase Fractionationed based EncyclopeDIA
관련글] Spectronaut의 direct DIA 분석 검증(Human plasma &Achilles tendon)
관련글] Spectronanut의 directDIA 분석과 Spectra library 기반의 정량분석 비교
관련글] Biognosys Spectronaut 의 Pulsa, directDIA를 이용한 DIA 검색능력 비교
관련글] Biognosys 사의 Spectronaut Pulsar의 DirectDIA 분석 (without Spectra library)
관련글] Biognosys 의 Spectronanut Pulsar 프로그램을 이용한 Spectra library 만들기
관련글] DIA Spectra library를 위한 Spin-column 형태의 hpRP, SCX, and HILIC 기반 펩타이드 분획
관련글] EncyclopeDIA, Spectronaut, PD2.4 with Overlap & non-Overlap DIA and DDA analysis
관련글] Overlap DIA 파일을 HTRMSConverter 로 변환하여 Spectronaut 분석하는 방법
관련글] EncyclopeDIA 을 이용한 DIA 단백질 정량분석
관련글] [Biognosys spectronaut] DIA 분석에서 Spectra library에 따른 단백질 검출차이
관련글] [Biognosys spectronaut] Configure condition 설정
관련글] Spectronaut 의 directDIA 분석과 Spectra library 기반의 정량분석 비교
관련글] Spectronaut 의 direct DIA 분석 검증(Human plasma &Achilles tendon)
관련글] [Biognosys spectronaut] DIA normalization Setting (Ovalbumin DIA 정량분석)
관련글] EncyclopeDIA, Spectronaut, PD2.4 with Overlap & non-Overlap DIA and DDA analysis
'프로테오믹스(단백체학)' 카테고리의 다른 글
| Chymotrypsin의 "Semi" cleavage 를 이용한 단백질 검출 I (0) | 2021.02.22 |
|---|---|
| Proteome Discoverer에서 Spectra library 기반 MSPepSearch로 단백질 검색하기 (0) | 2021.02.20 |
| Skyline의 DIA method 설정에서 Window 수와 Optimize window placement 에 따른 비교 분석 (QE-HF) (0) | 2021.02.18 |
| 적은수의 펩타이드를 정량분석에 있어서 PRM과 DIA 분석법 비교 (0) | 2021.02.17 |
| 10-Plex TMT로 Phosphopeptides 검출에 있어서 Sequest HT 와 MS Amanda 2.0 비교 (0) | 2021.02.14 |




댓글