앞에 글에서 EncyclopeDIA 분석법에 대해서 설명을 하였습니다. 동일한 시점에서 동일한 시료/장비를 이용하여 Overlap DIA, non-Overlap DIA, 그리고 DDA (LFQ)의 결과를 비교하였습니다.
Overlap DIA로 분석한 데이타는 EncyclopeDIA 와 Spectronaunt로 각각 분석하였습니다.
EncyclopeDIA로 분석시에는 GPF에서 얻어진 Spectra library를 사용하였고, Spectronaunt 분석시에는 HRMSconverter 를 이용하여 변환한뒤 DDA 로 얻어진 Spectra library를 사용하였습니다.
Non-overlap DIA의 경우 Spectronaunt 로만 분석하였습니다. 또한 DDA로 분석한 LFQ실험은 Proteome Discoverer 2.4로 분석하였습니다.
단백질 수 비교
DIA 분석에서의 비교분석한 결과는 아래의 그래프와 같습니다. Missing value 가 존재하는 단백질을 제외한 단백질의 수입니다. 전반적으로 유사한 단백질 수가 검출되었고 또한 높은 재현성을 보였습니다.
미비한 차이지만 단백질 수로 본다면 Overlap DIA+ EncyclopeDIA 분석법이 가장 많이 나왔습니다. Overlap DIA와 두 프로그램을 비교해본다면 Spectronaunt보다 EncyclopeDIA에서 단백질이 조금더 더 많이 검출되었습니다. 하지만 missing value는 Spectronanut가 더 적었습니다.
또한 Non-Overlap DIA를 Spectronanut로 검색했을시 Overlap DIA+EnclycopeDIA에서 검출된 수와 거의 유사한 수가 검출되었습니다. 아마 Spectronanut 프로그램은 non-Overlap DIA 분석법과 궁합이 잘맞는것 같습니다. 아니면 EncyclopeDIA 분석에 최적화된 Overlap DIA method가 Spectronaunt에 적합 하지 않을 수도 있습니다.

펩타이드 수 비교
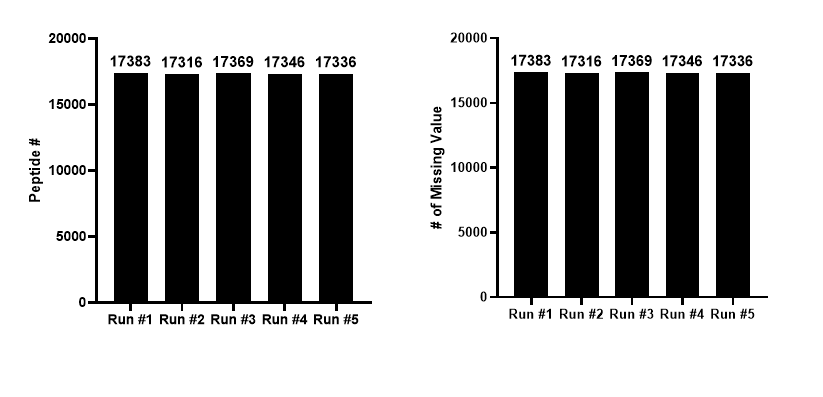
아래는 펩타이드에 대한 그래프입니다. 단백질에서의 결과와는 다르게 EncyclopeDIA+Overlap DIA에서 가장 낮은 수를 보였으나 Missing value는 가장 낮은 값을 보였습니다. EncyclopeDIA에서는 실제 시료와 동일한 LC 조건에서 GPF로 spectra library를 만듭니다. 즉 EncyclopeDIA 분석시 retention time 값을 고려하기 때문에 false-positive 비율이 아주 낮습니다. 반대로 여러조건에서 DDA 방식으로 얻어진 Spectra library를 사용하는 Spectra library의 경우 펩타이드와 매칭율이 높을 수도 있겠지만 false-positive 비율도 상대적로 높아지게 되는것 같습니다. 이러한 이유로 아래와 같은 결과가 나온것 같습니다.
DDA 분석
마지막으로 DDA 기반으로 얻어진 결과를 Proteome Discoverer 2.4의 Label-free quantitation을 수행하였습니다.
아래 그래프와 같이 전반적으로 단백질의 수는 앞의 DIA 분석법에 비해 약간 많이 검출되었습니다.

이 중에 한개의 unique peptide 검출된 단백질을 제거한 그래프는 아래와 같습니다. 위의 그래프와 비교해보면 숫자가 많이 줄었습니다. 한개의 펩타이드로 검출된 단백질이 상당히 많은것 같습니다. 이런 단백질의 경우 재현성과 정확성이 좋지 않을 가능성이 높습니다.

펩타이드에 대한 그래프는 아래와 같습니다.
DIA와 DDA를 직접 비교하기는 무리가 있을것 같습니다. DIA의 분석시 EncyclopeDIA와 Spectronaunt 경우 단백질(펩타이드)이 검출이 되더라도 단백질이 정량을 위한 값을 변환될때 조금더 까다로운 조건이 적용되는것 같습니다. (개인적인 생각). 정량분석에는 단백질의 수도 중요하지만 정확한 정량성과 재현성이 더 중요한것 같습니다.
관련글] DIA분석(QE)에 있어서 m/z 500-900 과 mz 400-1000 검색구간에 따른 결과 차이
관련글] Skyline의 DIA method 설정에서 Window 수와 Optimize window placement 에 따른 비교 분석 (QE-HF)
관련글] 적은수의 펩타이드를 정량분석에 있어서 PRM과 DIA 분석법 비교
관련글] PRM / DIA 정량분석법의 최적화를 위한 peak point 찾기 (실제 데이타)
관련글] PRM / DIA 정량분석법의 최적화를 위한 peak point 찾기
관련글] Biognosys Spectonaut directDIA vs Gas-phase Fractionationed based EncyclopeDIA
관련글] Spectronaunt 의 direct DIA 분석 검증(Human plasma &Achilles tendon)
관련글] Spectronanut 의 directDIA 분석과 Spectra library 기반의 정량분석 비교
관련글] Biognosys Spectronaunt 의 Pulsa, directDIA를 이용한 DIA 검색능력 비교
관련글] Biognosys 사의 Spectronaunt Pulsar의 DirectDIA 분석 (without Spectra library)
관련글] Biognosys 의 Spectronanut Pulsar 프로그램을 이용한 Spectra library 만들기
관련글] DIA Spectra library 를 위한 Spin-column 형태의 hpRP, SCX, and HILIC 기반 펩타이드 분획
관련글] EncyclopeDIA, Spectronaunt, PD2.4 with Overlap & non-Overlap DIA and DDA analysis




댓글