최근 논문과 강좌에서 많이 등장하고 있는 FAIMS를 드디어 테스트 해보게 되었습니다. 사실 장비에 뭔가 새로운것을 붙히는것을 그다지 좋아하지는 않습니다. 얻는것보다 잃는것이 많을 때가 있기 때문입니다. 업체들은 보통 장점만을 강조하며 또한 최적화된 상태에서 실험한 결과를 보여주기에 실제 실험실에서의 상황과 다를수 있기 때문입니다. 하지만 여러 논문에서 좋은 결과들이 나온것을 보면서 기대하는 마음으로 조금씩 테스트해보았습니다.

Nanoflow에서는 0 L/min을 사용을 해야 합니다. 그렇지 않으면 nanoflow에서는 gas로 인해 이온들이 질량분석기로 들어가는것을 방해받을수 있습니다. Resolution mode의 경우 대부분의 프로테오믹스 실험에서는 “standard resolution”을 사용합니다. “High resolution”의 경우 PRM과 같은 특정 펩타이드를 분석할시 m/z값이 매우 유사한 interferences가 존재할때 사용하면 좋습니다.
FAIMS는 특별히 최적화해야할 조건이 많이 없습니다. 서로 다른 CV값으로 최적화를 해야하지만 bottom-up proteomics에서는 기존의 문헌에서 최적화된 값이 -50V와 -70V를 동시에 사용하면 무난할것 같습니다. 무수히 많은 성질을 가지는 펩타이드가 존재하는 시료에서 모든 펩타이드를 만족하는 최적화된 값은 없기 때문입니다.
Base-Peak Chromatograms of BSA with Different FAIMS CVs
아래는 서로 다른 CV값에서 검출된 100fmol BSA의 Base-peak chromatograms 입니다.

문헌에서는 보긴하였지만 실제로 CV값에 따라 검출되는 피크들이 모양이 다르다는것을 확인하였습니다. 신기하기도 합니다. . -10 에서 -30V사에서는 거의 검출이 되지 않다가 -40V 부터는 서로 다른 패턴의 펩타이들이 검출이 되었습니다.
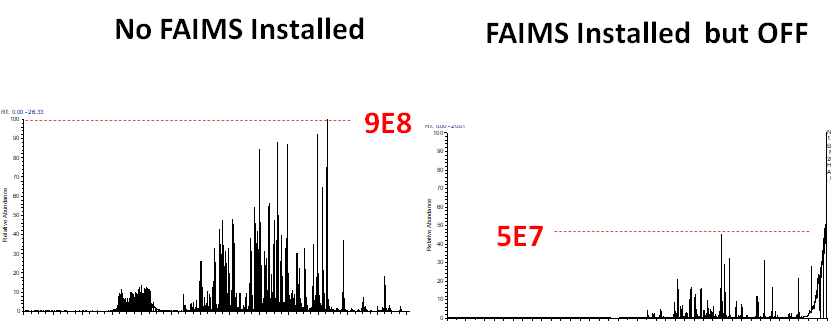
NO FAIMS-installed vs OFF FAIMS
FAIMS를 장착하지 않을때와 FAIMS를 장착하였지만 OFF 상태둔 두었을때의 BSA 스펙트럼입니다. FAIMS를 장착만 하였을때는 많은 이온들의 손실이 있는것을 알수 있습니다. 즉 voltage를 적용하지 않으면 이온들이 FAIMS를 통과하는 과정에서 손실이 된다는것을 알수 있습니다. 즉 voltage가 적용이 되어야지 펩타이드들이 FAIMS 안에서 효율적으로 나온다는것을 알수 있습니다.
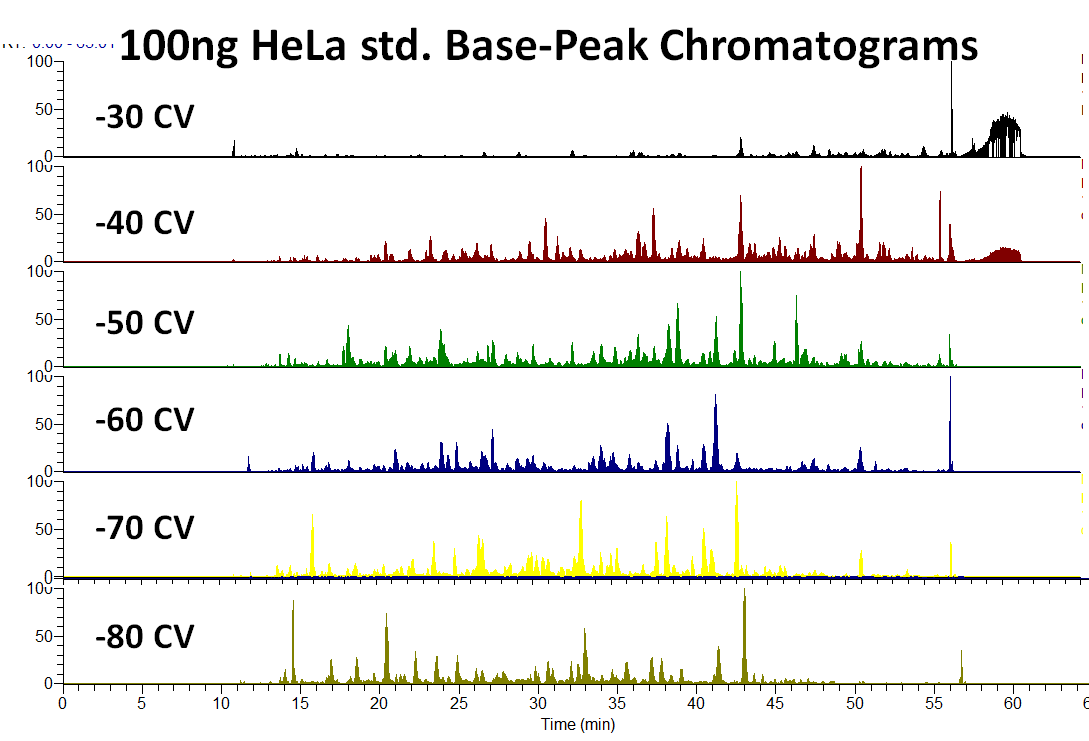
HeLa standard digest with different FAIMS CVs
100 ng HeLa standard digest를 서로 다른 CV값에서 측정한 크로마토그램입니다. BSA에서 얻어진 결과와 유사하게 서로 다른 형태의 크로마토그램을 보여줍니다.
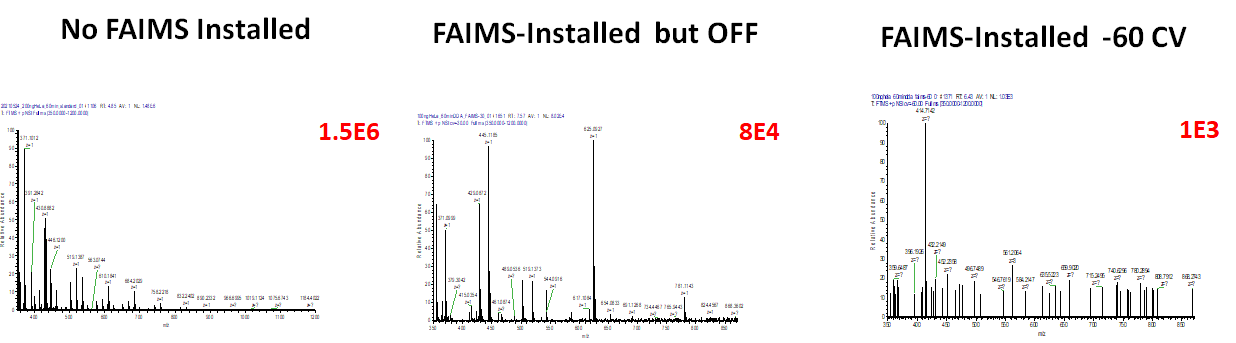
아래는 FAIMS를 장착하지 않았을때, 장착후 OFF, 그리고 -60V의 FAIMS 조건에서 보여지는 Background 스펙트럼입니다. FAIMS를 장착하지 않았을때는 일반적으로 보이는 형태의 스펙트럼이 보입니다. 그러나 FAIMS를 장착하였으나 OFF상태에서는 약간 다른 형태의 스펙트럼이 나 타났으면 피크 높이도 상당히 낮습니다. 특이한것은 -60V의 FAIMS조건에서는 background 피크가 거의 보이지 않습니다. 실제 펩타이드들은 정상적인 크로마토그램을 보여지는것을 감안하면 single charge를 가지는 background 피크들이 아주 효율적으로 제거되었음을 의미합니다
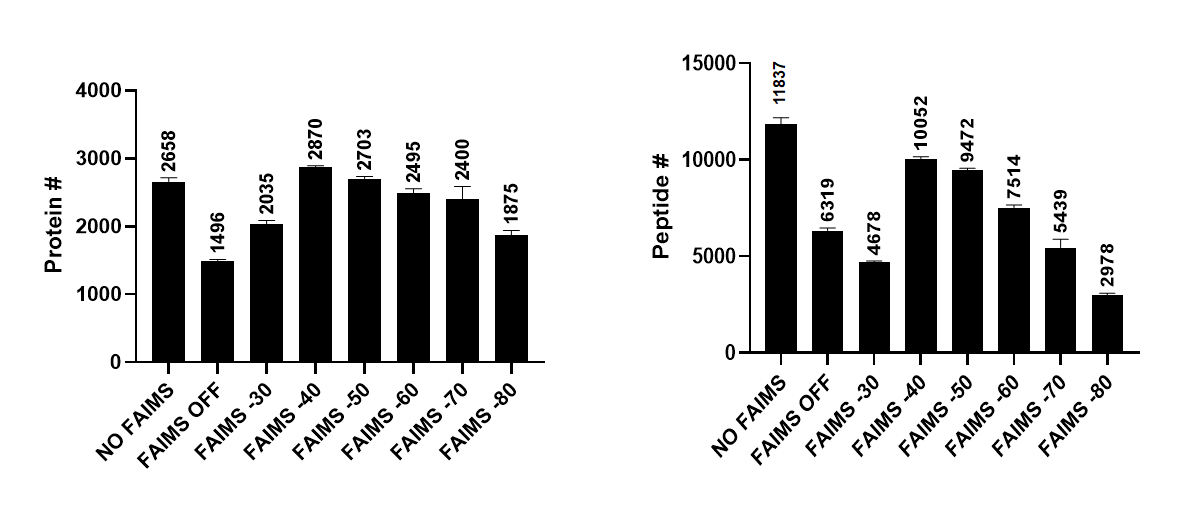
서로 다른 CV 값에서 검출된 단백질과 펩타이드의 수는 아래와 같습니다. FAIMS을 장착하지 않았을때의 결과가 대부분의 FAIMS CV값에서 얻어진것보다 좋습니다. 하지만 FAIMS -40V와 -50V에서 유사하거나 약간 높은 수의 단백질이 검출되었습니다. 그러나 펩타이드의 수는 FAIMS에서 훨씬더 적게 검출되었습니다. 기존의 문헌에서 설명된것 처럼 FAIMS는 CV값에 따라 펩타이드를 선택적으로 검출하기 때문입니다.
FAIMS의 경우 한개의 CV값으로 검출하는것 보다는 Dual값으로 동시에 검출할때 그 효과가 나타나게 됩니다. 서로 다른 CV값에서 서로 다른 펩타이드가 검출되기 때문입니다. Gas-Phase fractionation 의 효과를 볼수 있습니다. 그래서 두개의 서로 다른 CV값을 가지는 Dual method를 사용하여 결과를 얻었습니다. 이 Method를 만들기 위해서는 두개의 DDA experiment를 만들어야 됩니다. 두개의 experiment는 모두 동일하나 CV값만 다르게 설정하면 됩니다.
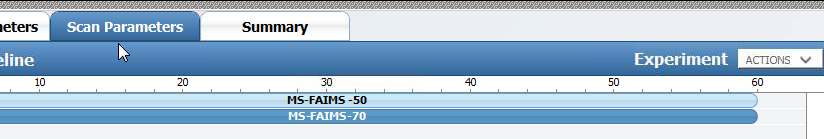
즉 한개의 FAIMS 값으로 Full MS 이후 MS2가 수행된후 다시 다른 CV값으로 Full MS를 수행한후 MS2가 수행됩니다. 두번의 DDA가 연속적으로 수행됩니다. 하지만 Exploris의 빠른 속도로 인해 두번의 DDA분석에서 손실되는 부분은 적은것 같습니다.
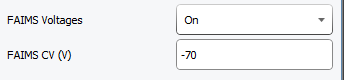
논문에서는 -50CV와 -70CV가 가장 좋은 조합이라고 설명합니다. 하지만 이번 실험에서는 -40CV에서 많은 펩타이드가 검출이 되었기에 -40CV와 조합을 만들었고 또한 -50CV도 포함시켰습니다.
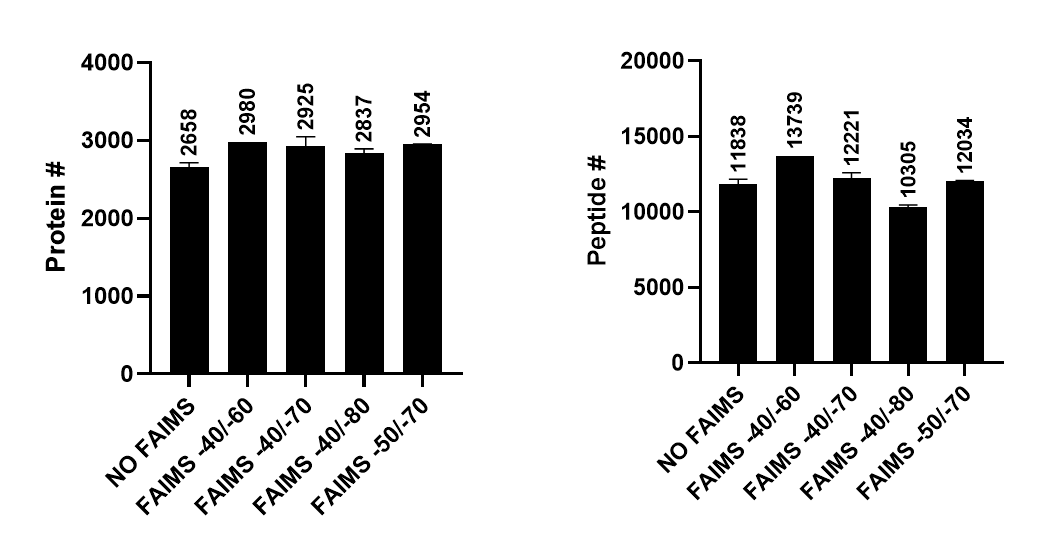
아래는 검출된 단백질과 펩타이드의 수를 비교한 그래프입니다. Dual로 했을때 검출된 단백질과 펩타이드의 수가 증가된것을 볼수 있습니다. 특별히 펩타이드의 수는 한개의 CV결과에 비해서 월등히 증가하였습니다.
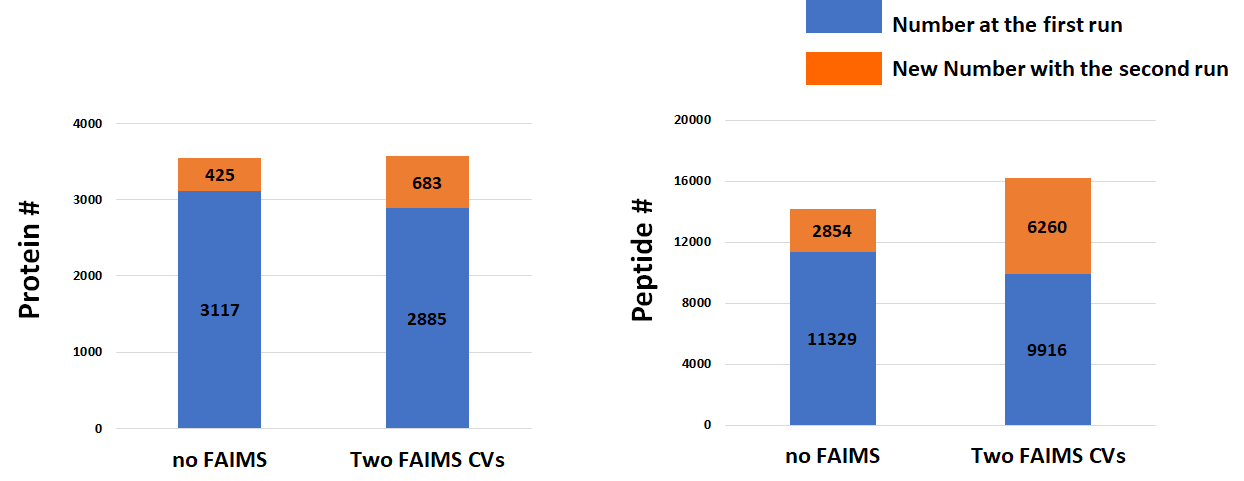
FAIMS를 장착하지 않은 상태에서 두 번 연속 분석하였을때와 한번의 분석이지만 Dual FAIMS CV로 분석하였을때
새롭게 검출되는 단백질과 펩타이드의 수를 비교해보 았습니다. 그래프에서 보듯이 서로 다른 CV에서 보다 다른 새로운 펩타이들이 검출이 되었습니다.
FAIMS를 설치하면서 가장 염려된 부분은 nanosource와 Entrance 사이에 설치된 FAIMS로 인해 이온들의 손실이 많지 않을까 하는것입니다. 또한 선택적으로 서로 다른 charge state의 펩타이드를 선별할수 있지만 이로 인해 또한 이온들의 손실이 있을수 있다고 생각했습니다. 이 테스트를 통해서 FAIMS가 다양한 연구에 적용될수 있는 가능성을 보았습니다.
관련글
Thermo Orbitrap Exploris 480 Benchmark (Data-Dependent Acquisition)
Thermo Orbitrap Exploris 480 Benchmark (Data-Dependent Acquisition)
2021년 초에 설치되었던 Exploris 480이 다른 큰 프로젝트를 수행하다가 드뎌 제 손으로 직접 테스트 해보게 되었습니다. 비록 완전 새것 상태에서는 바로 테스트를 못한것이 아쉬움이 있지만 그래
proteomicstechnology.tistory.com
Thermo Q-Exactive HF benchmark Experiments
Thermo Q-Exactive HF benchmark Experiments
오래전에 Refurbished QE-HF를 설치한후 Benchmark 실험을 한 데이타를 정리하여 보았습니다. 비록 완전 새것은 아니지만 특별히 장비에 문제가 없는 상태에서 구입을 하였습니다. 장비가 설치되면 Re
proteomicstechnology.tistory.com
Q-Exactive (Thermo Scientific) Benchmark data
Q-Exactive (Thermo Scientific) Benchmark data
저의 대학원생활의 첫 질량분석기는 지금은 볼수 없는 Agilent사의 iontrap이 었습니다. 그 당시 Thermo Finnigan 사의 LCQ 나 LTQ가 가장 좋은 질량분석기였습니다. 몇년후 LTQ를 구매하고 나서 Nature 급 논
proteomicstechnology.tistory.com
'프로테오믹스(단백체학)' 카테고리의 다른 글
| FAIMS가 장착된 Exploris 480에서 Cycle time (s), LC gradient와 CV값 에 따른 단백질 검출수 (0) | 2021.07.03 |
|---|---|
| Exploris 480 장비에서 Easy-IC를 이용한 internal calibration의 효율성 (0) | 2021.07.02 |
| Orbitrap Exploris 480 Benchmark (Data Independent Acquisition) (0) | 2021.06.29 |
| Thermo Orbitrap Exploris 480 Benchmark (Targeted Assay, PRM) (0) | 2021.06.29 |
| Thermo Orbitrap Exploris 480 Benchmark (Data-Dependent Acquisition) (0) | 2021.06.28 |




댓글