Orbitrap Eclipse를 사용하는데 있어서 기대감중 하나는 TMT 분석에 대한 성능이 향상되었다는것입니다. 기존의 Q-Exactive시리즈에서 사용했던 MS2 방법은 suppression 문제로 사용하지 않았었습니다. 대체방법으로 Orbitrap Fusion에서 MS3 SPS를 사용해서 이 문제를 많이 해결하였지만 단백질 검출수가 많이 떨어지는 단점이 있었습니다. 그래서 한동한 TMT 정량분석은 사용하지 않았고 DIA만을 사용하였습니다. DIA 분석법을 이용하면 비싼 TMT 를 사용하지 않고도 아주 좋은 결과를 얻었습니다. 하지만 여전히 TMT 분석을 선호하는 분들고 있고 실험전략에 따라 TMT분석이 더 효율적일수가 있습니다. 아마 Thermo Fisher에서 TMT 분석에 대한 광고를 잘 해와서 그런지 프로테오믹스를 잘 모르는 분들도 TMT 분석을 할수 있나고 물어봅니다.
TKO TMT 11-plex standard
현재 Thermo에서 실제 분석을 위한 TMT 11-plex standard가 판매되고 있습니다.
Pierce™ TMT11plex Yeast Digest Standard (thermofisher.com)

Yeast Proteome에서 상대적으로 높은 양으로 존재하는 3개의 단백질이 각각 deletion된 시료입니다.
Δ Met6 (P05694) : 126, 127N, 128N
Δ His4(P00815) : 128N, 128C, 129C
Δ Ura2Δ (P07259) : 129C, 130N, 130C
관련된 논문은 아래와 같습니다.


Thermo Fisher를 통해 1개를 주문하였습니다. 20µg Vial에 300불이 약간 넘습니다. 시료와 같이 온 설명지에는 40 µL 혹은 80 µL의 0.1% TFA / 5% Acetonitrile 로 녹이라고 되어 있습니다. 그러면 0.5 µg/µL 혹은 0.25 µg/µL가 됩니다. 하지만 Vial을 받아보면 아시겠지만 바닥의 표면적이 넓기 때문에 소량의 용매로 녹이다보면 시료 손실이 많이 날때가 있습니다. 즉 40µL로 녹인후 실제 tube에 옮기게 되면 40µL가 훨씬 못 미칩니다. Vial 표면과 Tip등에에 흡착되기 때문입니다. 그래서 100µL를 사용하여 200ng/uL로 만들었습니다.
MS3 SPS with Real-time Search
최근 논문에 발표된 대로 Orbitrap Eclipse에는 TMT 분석에 있어서 Real-time search 기능이 있습니다. 용어 그대로 분석중에 바로 검색이 진행됩니다. 진행속도는 약 5ms 정도가 됩니다. 검색은 Open source인 Comet search engine을 사용합니다. 기존의 MS3 SPS에서는 필요없이 MS3를 수행하거나 수행하더라도 Quality가 낮은 스펙트럼이 많이 생성되었습니다. 하지만 Real-time search를 통해 이러한 부분이 보완이 되었습니다. 이것은 크게 2가지 큰 유익이 있습니다.
하나는 MS2 스펙트럼의 실시간 검색을 통해 선택적으로 MS3를 수행합니다. Peptide-spectrum match(PSM)가 이루어진것만 MS3를 수행하게 됩니다. 다른 하나는 MS3를 수행하기 위한 선택하는 MS2 peak들을 선택적으로 정하게 됩니다. 즉 해당 펩타이드에 해당하는 MS2만을 선정하여 MS3를 넘어가게 됩니다. 속도와 정확도 모두 향상시키게 됩니다.
이번에는 Orbitrap Eclipse과 TKO TMT 11-plex standard를 이용하여 MS2, MS3 SPS, MS3 SPS+RTS 를 비교해보았습니다. 논문이나 회사의 설명서에 나온것만큼 실제 효과가 있는지 확인이 필요할듯 합니다.
Experimental workflow
아래 그림과 같이 진행하였습니다. 200 µg을 주입하였고 75µm ID x 15 cm 의 EasySpray 컬럼을 사용하였습니다.
질량분석기의 method는 Xcalibur에 원래 들어있는 방법을 변경하지 않고 사용했습니다.

MS3 SPS+RTS Method Parameters
아래는 전체적인 MS method입니다. MS2까지는 일반적인 방법으로 진행됩니다. MS2는 HCD fragmentation과 Iontrap detector를 사용합니다. 속도가 빠른 iontrap의 장점을 잘 살릴수 있습니다. 얻어진 MS2 스펙트럼을 이용하여 실시간으로 검색이 된후 MS3를 수행할것인지 수행한다면 어떤 peak들을 선택할지 결정합니다.

아래의 이미지에서 보듯이 MS2는 iontrap detector를 사용하되 속도를 높이기 위해서 Turbo scan rate를 사용합니다.
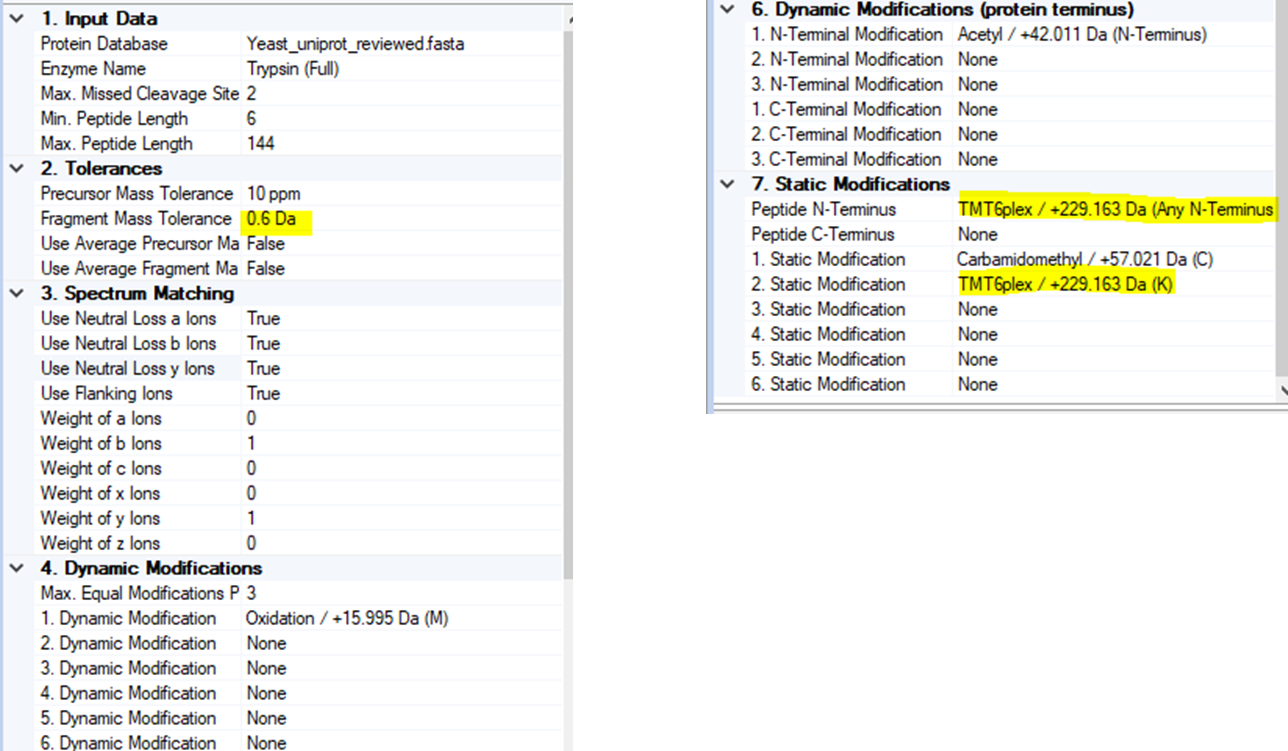
MS3에서는 SPS가 체크되었고 10개의 MS2 peak들을 모아서 MS3를 수행합니다. HCD 에너지는 65%로 상당히 높습니다. 어짜피 펩타이드 검출에 사용하는것이 아니라 reporter ion의 상대적인 높이를 측정하기 때문에 높은 값을 사용하였습니다. Scan range는 m/z 100 에서 500 입니다. 이것도 역시 Reporter ion들만 보기 위해서 좁은 영역을 사용하였습니니다. Real-time Seach는 Carbamidomethyl (C)와 TMT6plex(K, Nterm)이 포함되어 있습니다. 해당 fasta 파일을 넣어야 되지만 TKO TMT 11Plex standard 에 해당하는 fasta database는 method 자체에 포함되어 있습니다. 그외 여러가지 Parameter들이 있습니다. 일단 변경하지 않고 사용하였습니다.

Proteome Discoverer Database Search
각 조건마다 두번씩 분석한후 Proteome Discoverer 2.5로 분석하였습니다. Proteome discoverer에서의 분석에는 RTS가 포함되었는것과 포함되지 않는것에 구분이 없습니다. 하지만 MS3는 MS2와 검색방법이 약간 다릅니다. MS3 방법에서의 MS2는 iontrap으로 진행됨으로 tolerance가 높습니다 (여기서는 0.6 Da). MS3 스펙트럼으로는 펩타이드 검출이 아닌 reporter ion의 정량분석만 합니다.
Comparison of Protein and Peptide #
15 cm 컬럼으로 60 min gradient에서 얻어진 결과입니다. 아래 그래프에서 보듯이 MS2에 비해서 MS3에서 검출수가 줄어 듭니다. 이것은 아주 예상가능한 결과입니다. 하지만 놀라운것은 Real-time search를 사용한 MS3에서는 다른 두 방법에 비해서 검출수가 증가하였습니다. 앞에서 설명한 Real-time search의 기능으로 검출수가 증가된것입니다.

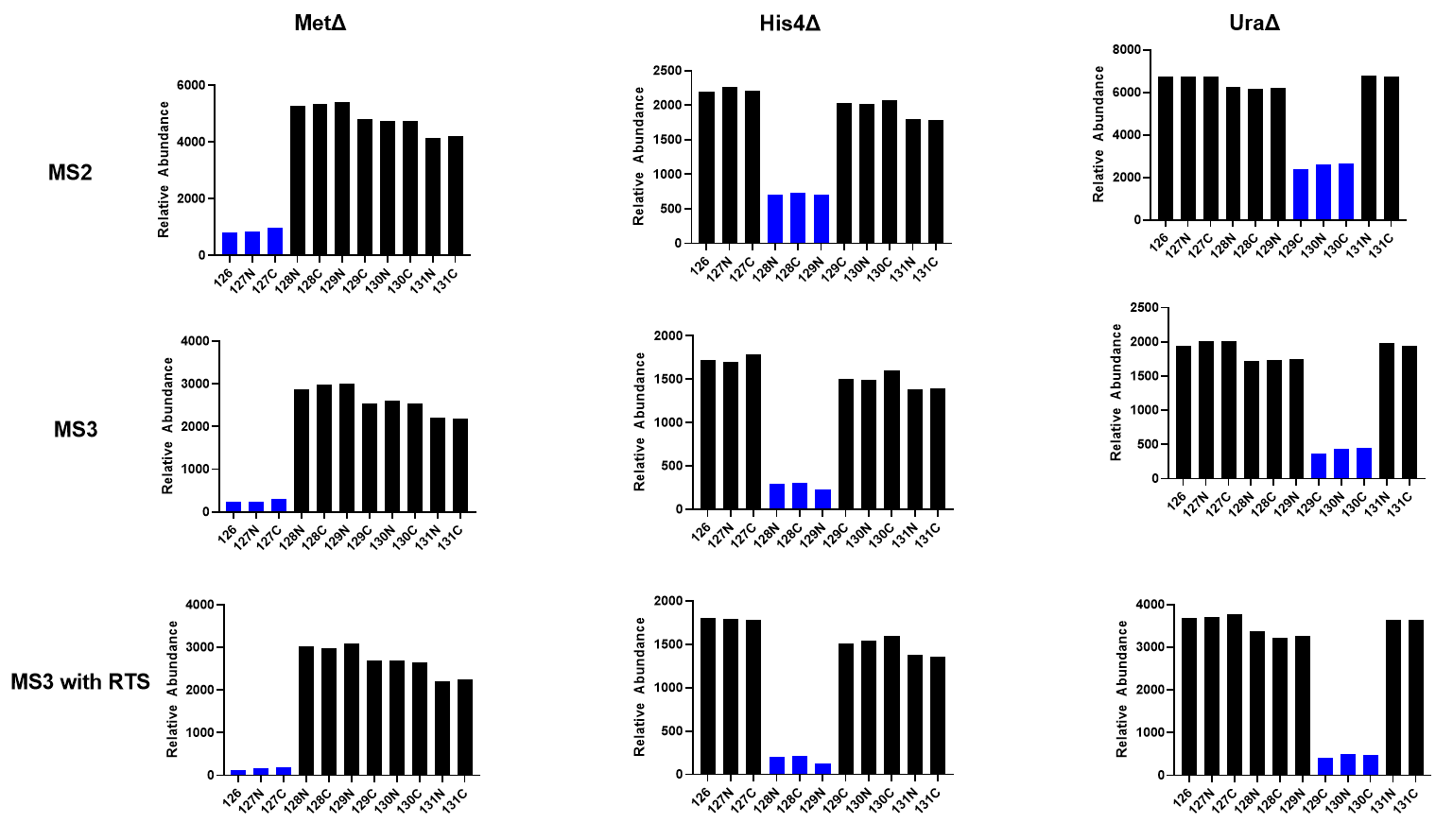
Relative Protein abundance of 3 Deletion Proteins
아래는 MS2, MS3 SPS, MS3 SPS with RTS에서 분석된 3개의 단백질에 대한 상대적인 정량그래프입니다.
회사에서 소개하는 결과에 비해서는 해당 단백질이 상대적으로 더 높은 비율로 검출이 된것 같습니다. 아마 사전 분획없이 짧은 LC gradient로 인해 Impurity peak들 포함된것일수도 있을듯 합니다. 하지만 MS2보다는 MS3가 더 이론적인 비율에 가까운것 같습니다. MS3 SPS 와 MS3 SPS + Real-Time Search를 비교해보면 Real-time Search가 포함된 분석에서 조금더 이론적인 값에 가까운것 같습니다. 아마 MS3에서도 여전히 impurity가 포함될수 있다고 해석할수 있습니다. 하지만 Real-time Search가 분석과정에서 이러한 impurity peak들을 제거하는데 도움이 되는것 같습니다.
Relative Abundance (MS2, MS3 SPS, MS3 SPS with RTS)
Comments
TKO TMT 11-plex Standard의 경우 극단적인 조건인것 같습니다. Deletion된 단백질이기 때문에 특별히 최적화하지 않아도 가시적으로 해당 단백질이 상당히 줄어든것을 볼수 있습니다. MS2 와 MS3 모두에서 상당히 줄어든것을 볼수 있습니다. 만약 5배 혹은 10배 정도의 차이를 가지는 단백질이 포함되어 있다면 보다 정밀하게 측정할수 있지 않을까 생각합니다. 문헌에 나와있는것과는 다르게 MS3 방법에서도 여전히 해당 단백질이 검출되었습니다. 아마 Impurity가 여전히 존재했던것 같습니다. 사전 분획을 한다면 이 부분도 약간은 해결되지 않을까 생각합니다. 하지만 소량밖에 없는 시료를 분획까지 하기는 아까울것 같네요.

'프로테오믹스(단백체학)' 카테고리의 다른 글
| Spectronaut 15 의 Direct DIA 테스트 (Plasma & HeLa) (0) | 2022.03.01 |
|---|---|
| Orbitrap Eclipse Data-Indepenant Acquisition-Cycle time vs Top N (0) | 2022.02.28 |
| Vanquish Neo/Orbitrap Eclipse 장비 QC를 위한 컬럼 (0) | 2022.02.23 |
| Vanquish Neo 에서 컬럼규격 설정 및 관련 셋팅방법 (0) | 2022.02.20 |
| Vanquish Neo nano UPLC 02 (3) | 2022.02.19 |




댓글