rASp-N을 이용하여 Sequence coverage 증가시키기
2018년 ASMS ( San Diego, California) 에서 우연히 Promega사의 Poster를 보았습니다. 새로나온 rAsp-N enzyme를 소개하는 내용이었습니다.
Asp-N digestion: Hydrolyzes Peptide Bonds on the N-Terminal Side of Aspartic and Cysteic Acid Residues (Asp and Cys)
종종 분석의뢰자가 분석하고자 하는 단백질의 sequence에 K, R 이 적절하게 존재하지 않아서 trypsin으로 충분한 정보를 얻지 못할때가 있습니다. 보통 Trypsin 대용으로 Gluc 혹은 Chymotrypsin을 사용하여 왔는데 이 rAsp-N도 분석서비스에 적용하면 좋을것 같았습니다. 학회를 돌아온후 얼마지나지 않아 주문하여 간단히 실험을 해보았습니다.
- Sample: Acetylated BSA (Sigma-Aldrich. B2518)
- Enzyme: rAsp-N (Promega, VA1160)
- Instrument: UltiMate™ 3000 RSLC coupled to Q-Exactive HFX
- DB search: Proteome Discoverer 2.3
아래 크로마토그램은 Trypsin (위)와 rAsp-N (아래) 으로 처리한 acetylated BSA의 Base-peak chromatogram입니다.
예상과 같이, 생성되는 peptide의 길이가 다르기 때문에 두 크로마토그램은 서로 다른 패턴으로 보여집니다.

얻어진 raw 파일을 Proteome discoverer 2.3에서 Asp-N "Full" 과 "Semi" 로 각각 검색하였습니다. 앞에서 설명한것과 유사하게 (바로가기), rAsp-N 에서도 "Semi" 로 검색시 더 높은 98% sequence coverage를 얻었습니다.

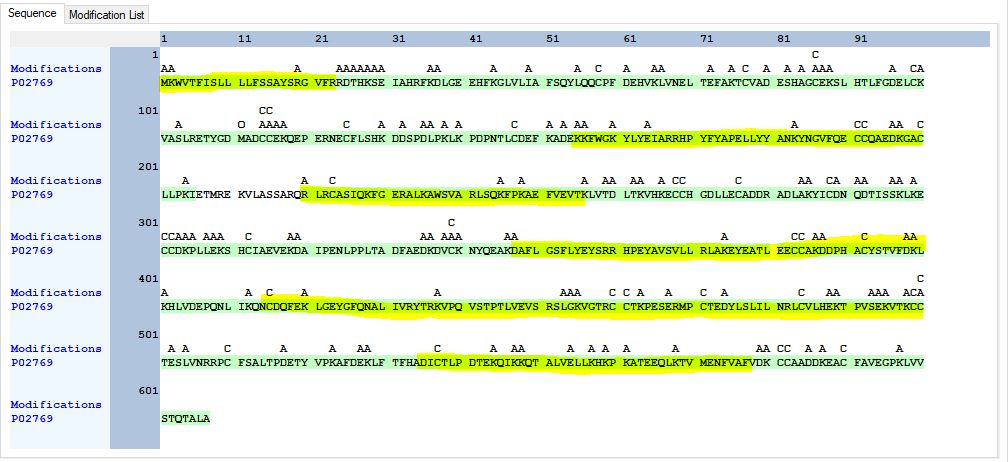
Trypsin에 비해서 고가이어서 자주 사용하지는 못할것 같지만 특정한 단백질 분석에 사용이 가능할것같다.
Proteinase K and Pepsin Digestion with BSA standard
Proteinase K and Pepsin Digestion with BSA standard
질량분석전 Protein digestion에서 Proteinase K 나 Pepsin 는 거의 사용하지 않습니다. 하지만 특정 펩타이드를 검출시 일반적인 enzyme (e.g, Trypsin. Lys-C, chymotrypsin등)이 적절치 않을때 사용될수 있습니..
proteomicstechnology.tistory.com
Chymotrypsin의 "Semi" cleavage 를 이용한 단백질 검출 II
Chymotrypsin의 "Semi" cleavage 를 이용한 단백질 검출 II
이전 글에 작성된 Chymotrypsin 처리후 데이타베이스 검색시 “Semi” digestion으로 검색하면 추가적인 펩타이드를 검출할수 있음을 설명하였습니다. 한번 더 확인차 BSA standard 를 이용하여 다시 실험
proteomicstechnology.tistory.com
Chymotrypsin의 "Semi" cleavage 를 이용한 단백질 검출 I
Promega사의 rAsp-N(P/N VA116A)의 사용후기 및 적용방법
데이타베이스 검색조건에서 Enzyme의 Semi 옵션으로 N-terminal 펩타이드 검출하는 법
데이타베이스 검색조건에서 Enzyme의 Semi 옵션으로 N-terminal 펩타이드 검출하는 법
Part1에서 소개한것에 이어 "Semi" 검색은 단일 단백질에서 추가적은 PTM sites를 찾는데도 유용하다. 간단히 acetylated BSA (Sigma-Aldrich, B2518)로 테스트해보았다 Sample: 250ng Acetylated BSA (on-column)..
proteomicstechnology.tistory.com
데이타베이스 검색조건에서 Enzyme의 Full 과 Semi 의 차이 알아보기
'프로테오믹스(단백체학)' 카테고리의 다른 글
| P-2000 Laser-Based Micropipette Puller 로 Nano column 만들기 (0) | 2020.04.06 |
|---|---|
| Trap-column 상태확인하는 방법 (Checking efficiency of trap-columns) (0) | 2020.04.05 |
| 데이타베이스 검색조건에서 Enzyme의 Semi 옵션으로 N-terminal 펩타이드 검출하는 법 (0) | 2020.04.05 |
| 데이타베이스 검색조건에서 Enzyme의 Full 과 Semi 의 차이 알아보기 (0) | 2020.04.05 |
| Morpheus (Free, Simple, Fast search tool) 사용법 알아보기 (0) | 2020.04.05 |




댓글